توقف مسیر اتصال پایانی غیر همولوگی (NHEJ)، توسط مهارکننده زیرواحد کاتالیزوری کیناز پروتئین وابسته به DNA (DNA-PKcs)
هپاتیت مزمن B، یک بیماری شدید کبدی است که توسط انتقال ویروس هپاتیت B (HBV) ایجاد می شود. DNA حلقوی بسته کووالانسی (cccDNA)، یک شکل استاندارد دوگانه فوق مارپیچ از ژنوم HBV، تعیین کننده اصلی پایداری ویروسی است. در حال حاضر، نوکلئاز های کریسپر/کَس 9 (CRISPR/Cas9) نشان داده شده اند تا DNA دو رشته ای را معرفی کنند که به HBV cccDNA شکسته می شود. آسیب تحمیلی، عمدتا در ترمیم نادرست cccDNA ، از اتصال پایانی غیرهمولوگی (NHEJ) ناشی می شود. NHEJ (اتصال پایانی غیرهمولوگی) پیشنهاد می شود تا فعالیت ضد HBV از کریسپر/کَس 9 (CRISPR/Cas9)را بهبود دهد و جهش cccDNA را افزایش دهد. در این تحقیق، ما فعالیت ضد HBV از کریسپر/کَس 9 (CRISPR/Cas9)و نتایج ترمیم cccDNA را در یک محیط NHEJ/HR اصلاح یافته، مورد ارزیابی قرار دادیم. NU7026، یک مهارکننده قوی NHEJ است که مانع از تخریب میانی کریسپر/کَس 9 (CRISPR/Cas9)از cccDNA شده است و منجر به حذف های تکرار شونده هدفمند شده است. ما نتیجه می گیریم که کریسپر/کَس 9 (CRISPR/Cas9)یک ابزار بسیار موثر برای تخریب cccDNA است و ابتدا نشان می دهد که مهار NHEJ باعث کاهش تخریب cccDNA می شود.
ویروس هپاتیت B (HBV)، به طور مداوم، 250 میلیون نفر را در سراسر جهان آلوده می کند، و بیش از یک میلیون نفر از عواقب هپاتیت مزمن B (CHB) می میرند، که عمدتا سیروز و کارسینوم هپاتوسلولار (HCC) است (3-1) . HBV متعلق به خانواده هپادناویریده است. ژنوم ویروسی ذره پوششی HBV شامل یک DNA دوگانه استاندارد جزئی سلولی است که از یک میانه RNA با استفاده از یک ترانس کریپتاز معکوس ، ترکیب می شود. پس از ورود، ویروس های HBV بدون پوشش هستند، و DNA حلقوی ساکن به نوکلئازی منتقل می شود که ان نوکلئاز از DNA حلقوی بسته کووالانسی (cccDNA) ترمیم می شود. cccDNA، مولفه کلیدی از چرخه عمر HBV است که بعنوان یک الگو برای رونویسی تمام RNAهای پیام رسان (mRNA) ویروسی شامل RNA پیش-ژنومی (pgRNA) بکار گرفته می شود (6-4).
توالی نسل-بعدی. نواحی هدف دار توسط کریسپر/کَس 9 (CRISPR/Cas9)و نواحی بدون هدف پتانسیل با استفاده از جفت پرایمرهای خاص و پلی مراز Q5 تقویت می شوند. امپلیکون ها با ژل تخلیص می شوند و با استفاده از کیت استخراج ژل کیاژن، استخراج می شوند، با یک فلورومتر کوبیت 2.0 (فناوری های عمر) اندازه گیری می شوند، و در نسبت های اکیومولار یکی می شوند. سپس، آداپتورها برای توالی ایلومینا پیوست می شوند. مجموعه اطلاعات (library) با 250 جفت بازخوانی نهایی با استفاده از ابزار MiSeq (ایلومینا) دنبال می شوند. نرم افزار FASTQC و نرم افزار جنیوس (Geneious ) برای ارزیابی کیفیت، همترازی منبع، دور انداختن بازخوانی های کیفیت پایین/ نوکلئوتیدها ، و محاسبه جایگزین ها و ایندل ها، استفاده می شوند. کیفیت توالی از قبل- باز و نمره های کیفیت پیش-توالی در تمام گروه ها بیشتر از 30 بودند.
Chronic hepatitis B is a severe liver disease caused by hepatitis B virus (HBV) infection. Covalently closed circular DNA (cccDNA), a super-spiralized, double-stranded form of the HBV genome, is the major determinant of viral persistence. CRISPR/Cas9 nucleases have been recently shown to introduce doublestranded DNA breaks into HBV cccDNA. The inficted damage results predominantly in erroneous repair of cccDNA by non-homologous end-joining (NHEJ). NHEJ has been suggested to enhance anti-HBV activity of CRISPR/Cas9 and increase cccDNA mutation. In this study, we assessed anti-HBV activity of CRISPR/Cas9 and cccDNA repair outcomes in an altered NHEJ/HR environment. NU7026, a strong inhibitor of NHEJ, prevented CRISPR/Cas9-mediated degradation of cccDNA and resulted in frequent on-target deletions. We conclude that CRISPR/Cas9 is a highly efective tool to degrade cccDNA and frst demonstrate that inhibiting NHEJ impairs cccDNA degradation.
Hepatitis B virus (HBV) chronically infects 250 million people worldwide, and more than a million people die from consequences of chronic hepatitis B (CHB), mainly cirrhosis and hepatocellular carcinoma (HCC)1–3 . HBV belongs to the family Hepadnaviridae. Te viral genome of the enveloped HBV particle comprises a circular, partially double-stranded DNA that is synthesized from an RNA intermediate using a reverse transcriptase. Upon entry, HBV virions are uncoated, and relaxed circular DNA is transported into the nucleus where it is repaired to form covalently closed circular DNA (cccDNA). cccDNA is the key component of that HBV life cycle that serves as a template for transcription of all viral mRNAs including pre-genomic RNA (pgRNA)4–6
Next-generation sequencing. Regions targeted by CRISPR/Cas9 and potential off- arget regions were amplifi d using pairs of specific primers and Q5 polymerase. Amplicons were gel-purifi d and extracted using Qiagen gel extraction kit, quantifi d with a Qubit 2.0 Fluorometer (Life Technologies), and pooled in equimolar ratios. Adapters for Illumina sequencing were then attached. Libraries were sequenced with 250 paired-end reads using the MiSeq instrument (Illumina). FASTQC sofware and Geneious sofware were used for quality assessment, reference alignment, discarding low-quality reads/nucleotides, and calculating substitutions and indels. Per-base sequence quality and per-sequence quality scores in all groups were >30.
نتایج
سمی بودن مولکول های کوچک و تاثیرات بر پایداری و چرخه سلولی
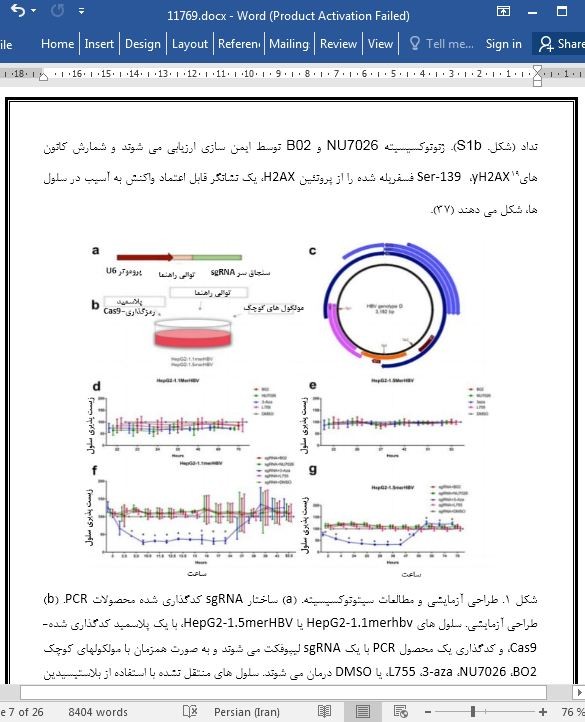
طراحی کریسپر/کَس 9 (CRISPR/Cas9).
فعالیت ضد HBV، کریسپر/کَس 9 (CRISPR/Cas9) در یک محیط NHEJ/HR.
مهار NHEJ توسط NU7026 منجر به ویرایش بیش از حد میانی-کریسپر/کَس 9 (CRISPR/Cas9) از HBV ccDNA می شود.
بحث
مواد و روش ها
کشت سلولی و ترانسفکشن
مولکول های کوچک
تحلیل قابلیت زیستی سلول
ساختارهای CRISPR/Cas9.
جداسازی اسیدهای نوکلئیک
تحلیل PCR.
ایمونوفلورسانس (پرتوافشانی ایمن).
تشخیص آپوپتوسیس
توالی نسل-بعدی
تحلیل آماری
منابع
Results
Toxicity of small molecules and efects on viability and cell cycle
Design of CRISPR/Cas9
Anti-HBV activity of CRISPR/Cas9 in an altered NHEJ/HR environment
Inhibition of NHEJ by NU7026 results in CRISPR/Cas9-mediated hyper-editing of HBV cccDNA
Discussion
Materials and Methods
Cell culture and transfection.
Small molecules
Cell viability analysis.
CRISPR/Cas9 constructs.
Isolation of nucleic acids
PCR analysis
Immunofuorescence
Apoptosis detection
Next-generation sequencing
Statistical analysis
References
- اصل مقاله انگلیسی با فرمت ورد (word) با قابلیت ویرایش
- ترجمه فارسی مقاله با فرمت ورد (word) با قابلیت ویرایش، بدون آرم سایت ای ترجمه
- ترجمه فارسی مقاله با فرمت pdf، بدون آرم سایت ای ترجمه