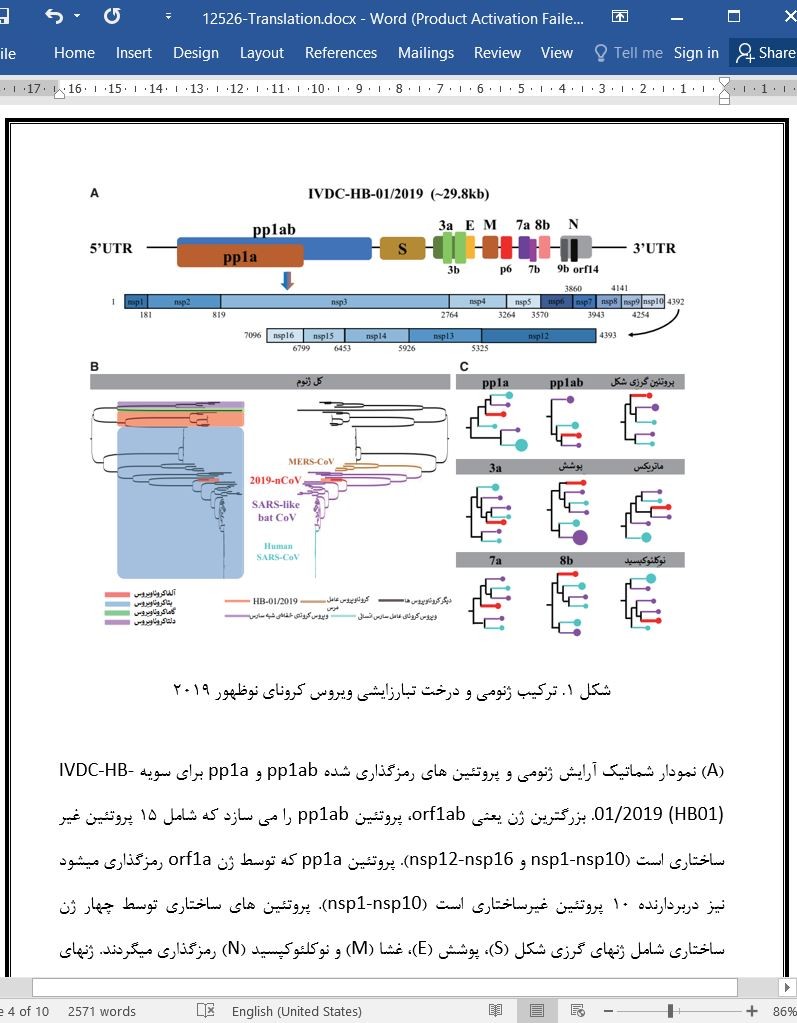
دانلود مقاله ترکیب ژنومی و واگرایی کروناویروس جدید (2019-nCoV) منشأگرفته از چین
گزارمانی جامع بر ژنوم ویروس کرونای تازه کشف شده (2019-nCoV) نمایانگر تفاوتی هایی میان 2019-nCoV و کرونا ویروس های عامل سندرم تنفسی حاد شدید (سارس) یا کروناویروس های شبه سارس بوده است. انجام مقایسه ای سازمان یافته به شناسایی 380 استخلاف آمینواسید در میان این کروناویروس ها انجامیده است که این امر ممکن است تنوع عملکردی و بیماری زایی ویروس کرونای نوظهور 2019 را موجب شده باشد.
ویروس کرونای (CoV) جدیدی که سازمان جهانی بهداشت به آن نام «ویروس کرونای نوظهور 2019» یا «2019-nCoV» را اطلاق کرده است، مسئول همه گیری اخیر ذات الریهای است که در اوایل دسامبر 2019 در شهر ووهان چین از ایالت هوبئی آغاز گردید . این همه گیری در ارتباط با بازار گسترده حیوانات و غذاهای دریایی بوده و تحقیقات به منظور تعیین منشأ عفونت در جریان است. تا به امروز، هزاران عفونت انسانی در چین و نیز بسیاری موارد سرایت در سراسر جهان تایید شده است .
ویروس های کرونا عمدتاً منجر به عفونت های تنفسی و دستگاه گوارشی می گردند و از نظر ژنتیکی به چهار سرده طبقه بندی میشوند: آلفا-کروناویروس، بتا-کروناویروس، گاماکروناویروس و دلتاکروناویروس . دو سرده ی اول عمدتاً پستانداران را آلوده می نمایند؛ درحالی که دو سرده ی گاما و دلتا غالباً پرندگان را آلوده می سازند . شش نوع از کروناویروس های انسانی پیشتر شناخته شده اند. این انواع عبارتند از HCoV-NL63 و HCoV-229E که متعلق به سرده آلفاکروناویروس ها می باشند؛ و HCoV-OC43 و HCoV-HKU1، ویروس کرونای عامل سندرم تنفسی حاد شدید (SARS-CoV) و ویروس کرونای عامل سندرم تنفسی خاورمیانهای (MERS-CoV) که به سرده بتاکروناویروس ها تعلق دارند . ویروس کرونا تا زمان همه گیری جهانی (پاندمی) سارس سال 2003، و پس از آن مرس 2012 و اخیراً همه گیری ویروس کرونای نوظهور 2019 توجه عمومی را به خود جلب نکرد . SARS-CoV و MERS-CoV شدیداً بیماری زا درنظر گرفته شده اند و احتمال بالایی وجود دارد که هردو از خفاش ها به زبادهای نخلی یا شترهای یک کوهانه و درنهایت به انسان ها انتقال یافته باشد .
An in-depth annotation of the newly discovered coronavirus (2019-nCoV) genome has revealed differences between 2019-nCoV and severe acute respiratory syndrome (SARS) or SARS-like coronaviruses. A systematic comparison identified 380 amino acid substitutions between these coronaviruses, which may have caused functional and pathogenic divergence of 2019-nCoV.
A novel coronavirus (CoV) named ‘‘2019 novel coronavirus’’ or ‘‘2019-nCoV’’ by the World Health Organization (WHO) is responsible for the recent pneumonia outbreak that started in early December, 2019 in Wuhan City, Hubei Province, China (Huang et al., 2020; Zhou et al., 2020; Zhu et al., 2020). This outbreak is associated with a large seafood and animal market, and investigations are ongoing to determine the origins of the infection. To date, thousands of human infections have been confirmed in China along with many exported cases across the globe (China CDC, 2020).
Coronaviruses mainly cause respiratory and gastrointestinal tract infections and are genetically classified into four major genera: Alphacoronavirus, Betacoronavirus, Gammacoronavirus, and Deltacoronavirus (Li, 2016). The former two genera primarily infect mammals, whereas the latter two predominantly infect birds (Tang et al., 2015). Six kinds of human CoVs have been previously identified. These include HCoV-NL63 and HCoV229E, which belong to the Alphacoronavirus genus; and HCoV-OC43, HCoVHKU1, severe acute respiratory syndrome coronavirus (SARS-CoV), and Middle East respiratory syndrome coronavirus (MERS-CoV), which belong to the Betacoronavirus genus (Tang et al., 2015). Coronaviruses did not attract worldwide attention until the 2003 SARS pandemic, followed by the 2012 MERS and, most recently, the 2019-nCoV outbreaks (China CDC, 2020; Song et al., 2019). SARS-CoV and MERS-CoV are considered highly pathogenic (Cui et al., 2019), and it is very likely that both SARS-CoV and MERS-CoV were transmitted from bats to palm civets (Guan et al., 2003) or dromedary camels (Drosten et al., 2014), and finally to humans (Cui et al., 2019).
- اصل مقاله انگلیسی با فرمت ورد (word) با قابلیت ویرایش
- ترجمه فارسی مقاله با فرمت ورد (word) با قابلیت ویرایش، بدون آرم سایت ای ترجمه
- ترجمه فارسی مقاله با فرمت pdf، بدون آرم سایت ای ترجمه