
دانلود رایگان مقاله ساختار ریبوزوم و مکانیسم ترجمه
انتشار ساختارهای بلوری از 50S و 30S زیرواحد ریبوزومی و ریبوزوم 70S دست نخورده، درک ما از سنتز پروتئین را متحول نموده است. این بررسی، تلاشی برای مرتبط نمودن ساختارها با داده های بیوشیمیایی و ژنتیکی برای شناسایی شکاف ها و محدودیت ها در دانش فعلی ما از مکانیسم های درگیر در ترجمه است.
مقدمه
در ترجمه، ترتیب و توالی کدون ها در mRNA، سنتز یک زنجیره پلی پپتیدی را هدایت می کند. این فرایند در ریبوزوم رخ می دهد، و حرکت tRNA و mRNA از طریق ریبوزوم، یک فرایند پیچیده است که ترکیبی از سرعت بالا با دقت بالا است. (بازنگری شده در Green and Noller, 1997). ریبوزوم، یک ذره ریبو-نوکلئوپروتئین بزرگ، شامل دو زیر واحد در تمام نمونه ها می شود. در باکتری ها، این زیر واحدها، 30S و 50S تعیین شده هستند، و با هم ریبوزوم 70S را تشکیل می دهند. هر زیر واحد ریبو دارای سه محل اتصال برای tRNA ، تعیین شده A (آمینو اسیل) است، که tRNA را آمینو اسیل ورودی می پذیرد. P (پپتیدیل)، که tRNA را با زنجیره پپتید در حال تولد نگه می دارد؛ و E (خروجی) که tRNA دی استیله شده را قبل از اینکه ریبوزوم را ترک کند، نگه می دارد. این زیر واحد 30S ، mRNA و حلقه-ساقه آنتی کدون tRNA را متصل می کند و با نظارت بر جفت شدن باز بین کدون و آنتی کدون در فرآیند رمزگشایی به راحت و کاربر پسند بودن ترجمه کمک می کند. این زیر واحد 50S، بازوهای پذیرنده tRNA را می بندد و تشکیل پیوند پپتیدی بین اسید آمینه های دریافتی در A-سایت tRNA و زنجیره پپتید در حال تولد متصل به P-سایت tRNA را کاتالیز می کند. هر دو زیرواحد در انتقال درگیر هستند که در آن tRNAها و mRNA دقیقا از طریق ریبوزوم، یک کدون در یک زمان، حرکت می کنند. ترجمه نه تنها ریبوزوم، بلکه عوامل پروتئین های اضافی را درگیر می کند که بسیاری از آنها GTPases فعال ریبوزوم می باشند. مطالعه ریبوزوم نیز به دلایل پزشکی مهم است، چرا که هدف بسیاری از آنتی بیوتیک ها است.
طرح کلی از ترجمه حدود چهار دهه پیش انجام شد، اما ثابت شده است که درک دقیق مکانیکی ترجمه به چند دلیل دست نیافتنی است. از آنجا که ریبوزوم برای تجزیه و تحلیل ساختاری با وضوح بالا بیش از حد بزرگ تا همین اواخر در نظر گرفته شد، و ابزار های بیوشیمیایی و ژنتیکی به صورت پیچیده همانند حال حاضر در دسترس نبود، پیشرفت کیفی به سمت درک درست از این مکانیسم دشوار بود. در نتیجه، بعد از عصر طلایی در دهه 1960 و 9170، ترجمه، به دیگر مسائل در بیولوژی مولکولی بعد پیوست.
هرچند، در دهه اخیر، پیشرفت سریعی ناشی از همگرایی رویکردهای مختلف صورت گرفته است. ابزارهای ژنتیکی و بیوشیمیایی خبره به طور فزاینده برای ترجمه اعمال شده اند. روند پیش از حالت پایدار، تشریح مراحل را در مسیر ترجمه میسر نموده است (Pape et al., 1998; Rodnina et al., 1997). (بازنگری شده در Agrawal and Frank, 1999; van Heel, 2000). ظهور تکنیک های بازسازی ذرات، همراه با میکروسکوپ cryoelectron (cryoEM) تصاویری را از ریبوزوم با افزایش جزئیات ارائه نمود. از آنجا که هیچ کریستالی مورد نیاز نیست و میزان مواد مورد نیاز در مقایسه با کریستالوگرافی کم است، این روش برای تعیین ساختار ریبوزوم در ترکیب با tRNA و mRNA مربوط به لیگاند و عوامل مختلف استفاده می شود (بازنگری شده در Agrawal و Frank, 1999; van Heeal,2000).
در نهایت، نزدیک به دو دهه پس از اولین گزارش از کریستالهای زیر واحد 50S (Yonath و همکاران، 1980)، پیشرفت های سریعی در بلورشناسی ریبوزوم صورت گرفت. این مورد نه تنها نیاز به کریستال های بهبود یافته از هر دو زیر واحد و کل ریبوزوم، بلکه نیاز به بر طرف شدن تعدادی از مشکلات کریستالوگرافی دارد (بررسی شده در Ramakrishnan and Moore, 2001). در نتیجه، ساختارهای اتمی هر دو زیرواحد و مدل مولکولی در 5.5 A از ریبوزوم 70S کلی اخیراً منتشر شده است. همراه با هم این ساختارهای کریستالی، میزان زیادی از اطلاعات را در مورد معماری کلی و جزئیات تعاملات پروتئین-RNA در هر زیرواحد و جزئیات تعامل ریبوزوم با لیگاندها مانند عوامل اولیه، mRNA و tRNA فراهم می کنند.
این بازنگری تلاش می کند تا داده های بیوشیمیایی اخیر و کلاسیک را در پرتوی ساختارها و در زمینه مسیر ترجمه بازبینی نماید. همانطور که توسط ماهیت حفاظت شده قابل توجه نواحی حیاتی ریبوزوم و ساختار کلی tRNAها قضاوت شده است، بسیاری از فرآیندهای مرکزی مانند شناخت tRNA، تشکیل پیوند پپتید و تغییر موقعیت ممکن، احتمالاً در طول عمر باید یکسان باشند. هرچند، ترجمه یوکاریوتی، پیچیده تر است و فراتر از آن تحت تنظیمات است. در این بازنگری، ما به طور کلی روی ترجمه در باکتری ها تمرکز می کنیم که در آن بیشتر اطلاعات بیوشیمیایی از کار استفاده از E.coli می آید و ما از شماره گذاری E.coli برای باقیمانده ها استفاده می کنیم مگر اینکه بیان شده باشد.
ساختارهای کریستالی ریبوزوم
مشخص کردن ساختار های مختلف کریستال ریبوزومی که همراه با محدودیت های آن مشخص شده است، در ابتدا می تواند مفید باشد. اول از همه ساختار ریبوزوم با وضوح بالا که باید منتشر شود، ساختار 2.4زیر واحد 50S از آرکئن Haloarcula marismortuii (Ban و همکاران، 2000) بود. این ساختار شامل بسیاری از زیر واحدهای 50S، از جمله تمام مرکز های مهم ترانسفراز پپتیدیل، اما در وضوح بالا، ویژگی های برجسته مانند ساقه L1، منطقه L11-RNA، و ساقه L7/L12می شود که همه آنها از نظر عملکردی در عوامل اتصال و انتقال که مختل شده به نظر می رسند، مهم هستند. برخی از حلقه های بنیادی RNA که در ارتباط با زیر واحد 30S دخیل هستند نیز بی نظم می باشند. این زیر واحد 50S Haloarcula تنها ساختار تا به امروز با وضوح کافی برای دیدن مولکول های آب، یون های فلزی، و تغییرات پایه با هر درجه از یقین است. به احتمال زیاد آنها برای درک ما از تا شدن و پایداری RNA حیاتی هستند و می توانند نقش هایی حیاتی را بازی کنند.اخیراً، ساختار زیرواحد 50S در تفکیک 3.1A از یک باکتری مزوفیلیک Deinococcus radioduarans گزارش شده است (Harms et al., 2001). RNA در این 50S دارای تطابق بسیار مشابه با RNA گزارش شده اصلی برای Haloarcula 50S, است، اما این ساختار شامل نواحی می شود که در Haloarcula 50S مختل شده اند، مانند L1 stalk, the L11/ ناحیه RNA و برخی از حلقه های بنیادی RNA که پل هایی را به زیرواحد 30S می زند.
دو ساختار مستقل از زیرواحد 30S از باکتری Thermus thermophilus، یکی در 3.3 A از یک گروه در موسسات Max Planck/Weizmann (Schluen zen et al., 2000)) و دیگری در 3.05 A از یک گروه در MRC (Wimberly et al., 2000) در آخر سال گزارش شد. تفاوت ها بین دو ساختار در جایی دیگر مورد بررسی قرار گرفته است (Ramakrishnan and Moore, 2001). به طور خلاصه، ساختار MRC نشاندهنده یک مدل اتمی کامل ضروری زیرواحد 30S است و تعدادی از تفاوت های چشمگیر در تفسیر RNA و اجزای پروتئین بین دو ساختار وجود دارد. هرچند، ساختارهای اخیر از گروه Max Planck/Weizmann (Pioletti et al., 2001) در توافق خوبی با ساختار MRC قرار دارند، به طور اورجینال منتشر شده اند.
The publication of crystal structures of the 50S and 30S ribosomal subunits and the intact 70S ribosome is revolutionizing our understanding of protein synthesis. This review is an attempt to correlate the structures with biochemical and genetic data to identify the gaps and limits in our current knowledge of the mechanisms involved in translation.
Introduction
In translation, the sequence of codons on mRNA directs the synthesis of a polypeptide chain. This process takes place on the ribosome, and the movement of tRNA and mRNA through the ribosome is a complicated process that combines high speed with high accuracy (reviewed in Green and Noller, 1997). The ribosome, a large ribonucleoprotein particle, consists of two subunits in all species. In bacteria, the subunits are designated 30S and 50S, and together make up the 70S ribosome. Each subunit has three binding sites for tRNA, designated the A (aminoacyl), which accepts the incoming aminoacylated tRNA; P (peptidyl), which hold the tRNA with the nascent peptide chain; and E (exit), which holds the deacylated tRNA before it leaves the ribosome. The 30S subunit binds mRNA and the anticodon stem-loops of tRNA, and contributes to the fidelity of translation by monitoring base pairing between codon and anticodon in the decoding process. The 50S subunit binds the acceptor arms of tRNA and catalyzes peptide bond formation between the incoming amino acid on A-site tRNA and the nascent peptide chain attached to the P-site tRNA. Both subunits are involved in translocation, in which the tRNAs and mRNA move precisely through the ribosome, one codon at a time. Translation involves not only the ribosome, but additional protein factors, many of which are GTPases activated by the ribosome. The study of ribosomes is also important for medical reasons, since it is the target of many important antibiotics.
The overall scheme of translation was determined about four decades ago, but a detailed mechanistic understanding of translation has proved elusive for several reasons. Because ribosomes were considered too large for high-resolution structural analysis until recently, and biochemical and genetic tools were not as sophisticated as they are now, qualitative progress toward an understanding of mechanisms proved difficult. As a result, translation took a back seat to other problems in molecular biology after its golden age in the 1960s and 70s.
In the last decade, however, rapid progress has been made due to a convergence of various approaches. Increasingly sophisticated biochemical and genetic tools have been applied to translation. Pre-steady-state kinetics has allowed the dissection of steps in the translation pathway Pape et al. 1998, Rodnina et al. 1997. The advent of single particle reconstruction techniques, combined with cryoelectron microscopy (cryoEM) began to provide images of ribosomes in increasing detail. Because no crystals are required and the amount of material required is minuscule compared to crystallography, this technique has been used to determine the structure of ribosomes in complex with tRNA and mRNA ligands and various factors (reviewed in Agrawal and Frank 1999, van Heel 2000).
Finally, nearly two decades after the first report of crystals of 50S subunits (Yonath et al., 1980), rapid progress has been made in the crystallography of ribosomes. This required not only improved crystals of both subunits and the whole ribosome, but also that a number of crystallographic difficulties be overcome (reviewed in Ramakrishnan and Moore, 2001). As a result, atomic structures of both subunits and a molecular model at 5.5 Å of the entire 70S ribosome have been published recently. Together, these crystal structures provide a tremendous amount of information on the global architecture and details of protein-RNA interactions in each subunit, as well as details of the interaction of the ribosome with ligands such as initiation factors, mRNA, and tRNA.
This review is an attempt to revisit both classical and recent biochemical data in light of the structures and in the context of the translation pathway. As judged by the remarkably conserved nature of critical regions of the ribosome and the universal structure of tRNAs, many of the central processes such as tRNA recognition, peptide bond formation, and possibly translocation, are likely to be the same across all kingdoms of life. However, eukaryotic translation is more complex and far more subject to regulation. In this review, we focus entirely on translation in bacteria, where most of the biochemical information comes from work using E. coli, and we use E. coli numbering for residues unless otherwise stated.
Crystal Structures of the Ribosome
It would be useful at the outset to specify the various ribosomal crystal structures that have been determined, along with their limitations. The first of the high-resolution ribosome structures to be published was the 2.4 Å structure of the 50S subunit from the archaean Haloarcula marismortuii (Ban et al., 2000). The structure comprises most of the 50S subunit, including all of the important peptidyl transferase center, but at high resolution, prominent features such as the L1 stalk, the L11-RNA region, and the L7/L12 stalk, all of which are functionally important in factor binding and translocation, appear to be disordered. Some stem-loops of RNA that are involved in contacts with the 30S subunit are also disordered. The Haloarcula 50S subunit is also the only structure to date with sufficient resolution to see water molecules, metal ions, and base modifications with any degree of certainty. They are likely to be crucial to our understanding of the folding and stability of RNA and may also play important functional roles. Recently, the structure of a 50S subunit at 3.1 Å resolution from a mesophilic bacterium Deinococcus radioduarans has been reported (Harms et al., 2001). The RNA in this 50S has a very similar conformation to that reported originally for the Haloarcula 50S, but the structure includes some of the regions that were disordered in the Haloarcula 50S structure, such as the L1 stalk, the L11/RNA region, and some of the RNA stem loops that make bridges to the 30S subunit.
Two independent structures of the 30S subunit from the bacterium Thermus thermophilus, one at 3.3 Å from a group at the Max Planck/Weizmann Institutes (Schluenzen et al., 2000) and another at 3.05 Å from a group at the MRC (Wimberly et al., 2000), were reported last year. Differences between the two structures have been discussed elsewhere (Ramakrishnan and Moore, 2001). Briefly, the MRC structure represents an essentially complete atomic model of the 30S subunit, and there are a number of significant differences in interpretation of both the RNA and protein components between the two structures. However, more recent structures from the Max Planck/Weizmann group (Pioletti et al., 2001) are in good agreement with the MRC structure originally published.
مقدمه
ساختارهای کریستالی ریبوزوم
راه اندازی
مروری کلی بر چرخه طویل شدن
کدگشایی
نقش EF-Tu
ترانسفراز پپتیدیل
جابجاشدگی
مدل هایی برای جابجاشدگی
نقش EF-G
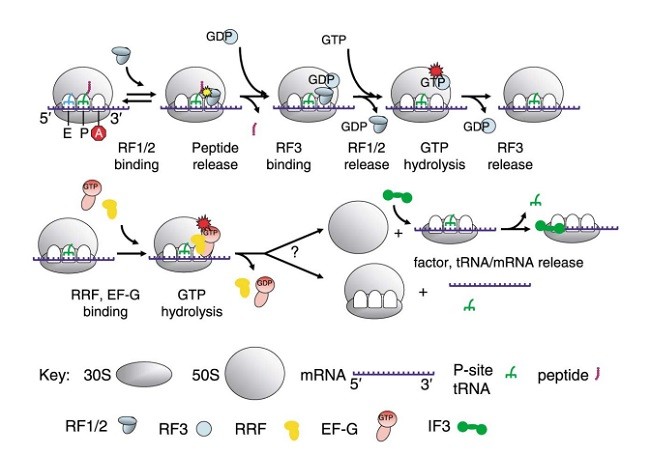
خاتمه
عامل بازيافتي ريبوزوم
نتايج
Introduction
Crystal Structures of the Ribosome
Initiation
Overview of the Elongation Cycle
Decoding
The Role of EF-Tu
Peptidyl Transferase
Translocation
Models for Translocation
The Role of EF-G
Termination
Ribosome Recycling Factor
Conclusions
Acknowledgements
References
