
دانلود رایگان مقاله تاریخچه ویرایش ژنوم در پستانداران
چکیده
ویرایش ژنوم در حال حاضر یک روال معمول در بسیاری از آزمایشگاه های ژنتیک پستانداران است. سابقه به ظاهر کوتاه اما قوی دستورالعمل های ویرایش ژنوم نشان می دهد که چگونه یک تکنولوژی مخرب می تواند به طور جهانی در یک زمینه مورد استفاده قرار گیرد ، زمانی که دریافتند که این روش جدید می تواند ژنوم پستاندار را در مکان های خاص تغییر دهد،به نتایج کارآمد و قوی دست یافتند . این بررسی خلاصه ای از توسعه اولیه ویرایش ژنوم با استفاده از نوکلئاز ها ، از آزمایش های اولیه ای که از مگانوکلئاز های مخمر استفاده کردند ، تا آخرین نوکلئازهای پروکاریوتی که برای دستکاری ژنوم دقیق استفاده می شوند، ارائه می دهد . نوکلئازهای ویرایش ژن متعلق به یکی از سه دسته شناخته شده زیر هستند:
1. zinc-finger nucleases (ZFN) : نوکلئاز انگشت روی
2. (TALEN) transcription activator-like effector nucleases: افکتور نوکلئاز شبه فعال کننده رونویسی
3. clustered regularly interspaced short palindromic repeats (CRISPR) and their associated proteins (Cas) tools : تناوبهایِ کوتاهِ پالیندرومِ فاصلهدارِ منظمِ خوشهای و پروتئین های مرتبط با آن
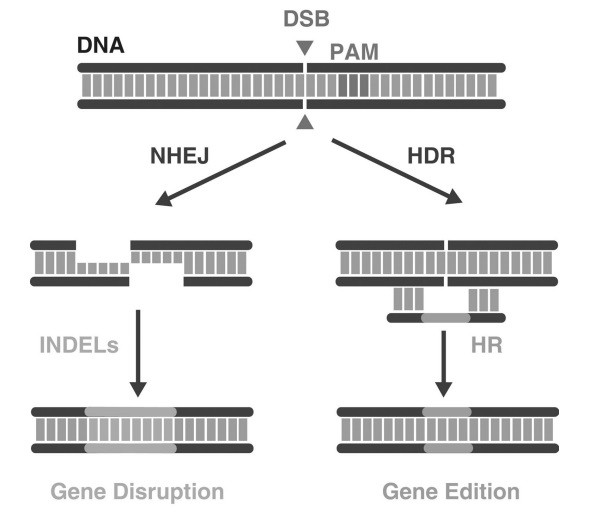
همه این سه روش بر اساس یک اصل عمل می کنند؛ همه آنها قادر به ایجاد یک شکست دو رشته (DSB) در توالی مشخصی از ژنوم هستند که سپس این شکست با مکانیسم های اندوژن ترمیم DNA اصلاح می شود. DSB ها می توانند از طریق اتصال انتهاهای غیر همولوگ (NHEJ) ترمیم شوند، و در نتیجه منجر به حذف و یا جایگزینی های کوچک (INDEL ها) در ژنوم می شود و از اینرو اغلب منجر به اختلال در ژن می شوند. به جای آن، DSB ها می توانند با استفاده از مکانیسم ترمیم بر اساس همولوژی (HDR)، در حضور توالی های DNA همولوگوس دهنده ، ترمیم شوند و منجر به رویدادهای ویرایش ژن شوند.
مقدمه
تبادل توالی ژنومی اندوژنیک برای مولکول های DNA اهداکننده خارجی با استفاده از نوترکیبی همولوگ (HR) فرایندی است که برای دهه ها شناخته شده است. مطالعات اولیه توسط مرحوم الیور اسمیتیس نشان داد که چگونه مولکولهای همولوگ DNA میتوانند دوباره با هم ترکیب شوند و به درستی در مکانهای کروموزومی تعریف شده در ژنوم پستانداران جایگزین شوند (Smithies et al.، 1984، 1985). این یافته ها برای پیش بینی و توسعه روش های هدف گیری ژن در سلول های بنیادی جنینی موش (ES) موثر بود و Smithies همراه با ماریو Capecchi و مارتین ایوانز به طور مشترک جایزه نوبل در فیزیولوژی و پزشکی در سال 2007 (مجله 2007) را به خاطر این کشف دریافت کردند. در این آزمایشات اولیه ، بازده حفظ یکپارچگی ژنی که هدف HR قرار می گیرد در رده های سلولی کشت داده شده، حتی در حضور انتخاب، نسبتا کم بود (در بهترین حالت 1 در 1000) (Smithies و همکاران 1985) .با این وجود، این روش حداقل 10 برابر کارآیی بیشتری نسبت به تلاش برای قرار دادن یک قطعه ژنتیکی DNA در محل کروموزوم مربوط به آن توسط HR با استفاده از میکرواینجکشن مستقیم پیش هسته ای تخمک های بارور شده موش دارد ؛ همانطور که با تلاش های رالف برینستر و همکاران نشان داده شد. (Brinster et al. 1989).
به سرعت واضح شد که باز کردن مولکول DNA اهدا کننده از طریق شکست دو رشته (DSB) در منطقه همولولوژی بطور قابل توجهی فرکانس HR درون ژن را افزایش می دهد (Kucherlapati و همکاران 1984؛ Smithies و همکاران 1985). زمانی که آلان برادلی افزایش بهره وری هدف گیری را با استفاده از وکتورهای insertion در مقابل replacement نشان داد؛ مشاهدات مشابهی چند سال بعد در سلول های موشی ES تأیید شد، (Hasty et al.، 1991). بنابراین، مزایای کارایی ادغام برای آنهایی که با القای DSB ها در الگوی DNA اهدا کننده به هم متصل می شوند، بیشتر است ، بنابراین در معرض قرارگیری توالی های همولوگ در پایان این مولکول های DNA، یک مفهوم کلی بود که به طور کامل نهادینه شده بود. مشاهدات نقش اساسی در تحولات بعدی ایفا می کند که منجر به اولین راهبردهای ویرایش ژنوم می شود.
در واقع، گام بعدی در راستای ایجاد اولین راهبردهای ویرایش ژنوم براساس استفاده از مگانوکلئازهای I-SceI مخمر بود که یک اندونوکلئاز برش دهنده نادر بود که توالی مورد شناسایی آن 18 نوکلئوتید طول داشت و مسئول جایگزینی اینترون در میتوکندری های مخمر بود (Jacquier and Dujon 1985 ). 10 سال پس از کشف آن، نشان داده شده است که مگانوکلئاز I-SceI می تواند برای القای HR در کروموزوم های پستانداران با فراوانی دو مرتبه بیشتر از HR خودبخودی (Choulika et al.، 1995) مورد استفاده قرار گیرد. نویسندگان نشان دادند که ترمیم موثر DSB های خاص القا شده توسط I-SceI با مولکولهای DNA اهداکننده حاوی مناطق همولوگ در کنار سایت I-SceI اندوژن در سلول های موشی صورت می گیرد که احتمالا یکی از اولین تلاش ها برای استفاده از استراتژی های ویرایش ژنوم بر اساس I -SceI meganuclease برای ایجاد DSB برای ترمیم با مکانیسم های سلولی اندوژن بود. پس از آن، معرفی رستریکشن سایت های I-SceI در لوکوس ژنومی موش توسط HR در سلول های ES و بیان موفقیت آمیز این مگانوکلئاز در حضور یک وکتور جایگزین با توالی های homologous DNA در اطراف سایت I-SceI معرفی شده، منجر به افزایش قابل توجه کارایی هدفگیری شد (تحریک 100 برابر) (Cohen-Tannoudji و همکاران، 1998).البته، این روش طولانی و پر زحمت بود و به دو مرحله متوالی HR و دو نشانگر انتخابی نیاز داشت. همچین خیلی عملی نبود، زیرا القای ادغام مبتنی بر I-SceI به شدت وابسته به معرفی قبلی سایت های مورد شناسایی I-SceI در لوکوسی بود که باید اصلاح می شد. علاوه بر این، گزارش های اخیر نشان می دهد که HR با مگانوکلئازهای القا شده توسط DSB ها ممکن است در سلول های پستاندار وابسته به لوکوس باشد (Fenina et al.، 2012). معرفی سایت های مگانوکلئاز I-SceI در هر دو انتهای ترانس ژن ها و تزریق همزمان با مگانوکلئاز همچنین باعث افزایش کارایی ترانسژنز در جنین های medaka fish (Thermes et al. 2002) و زنوپوس (Pan et al. 2006), اما نه در جنین های پستانداران شد. به تازگی، بر این محدودیت با استفاده از یک مگانوکلئاز اصلاح شده با سیگنال لوکالایز هسته ای (NLS) غلبه شده است، که به طور موثر ترانس ژنزیس بین جنین های موش و خوک را واسطه گری می کند (وانگ و همکاران 2014).
محدودیت آشکار توالی هدف مرتبط با I-SceI در نهایت با تولید مگانوکلئازهای مهندسی شده با سایت های شناسایی سفارشی(تجاری شده) ، هنگامی که آنها بصورت تجاری در دسترس قرار گرفتند ، برطرف شد. یکی از این مگانوکلئازهای مهندسی شده، که از I-CreI استخراج شده بود، برای ایجاد نوترکیبی هدفمند با فرکانس بالا در ژن SCID در سلول های انسانی مورد استفاده قرار گرفت (Grizot et al.، 2009). اخیرا، یکی دیگر از I-CreI مهندسی شده، که یک توالی 22 جفت بازی از ژن Rag1 را برش می دهد، برای نشان دادن رویدادهای موثر هدفمند در جنین های موش و رت (Menoret و همکاران 2013) مورد استفاده قرار گرفت.
به رسمیت شناختن اینکه مگانوکلئازها جز اولین مواردی بودند که به قصد ویرایش ژنوم مورد استفاده قرار گرفتند، و قبل از سایر نوکلئازهای مشهور و جدید (nucleases of zinc finger، ZFN؛ افکتورهای مرتبط با پروتئین فعال کننده رونویسی، TALEN؛ پروتئین های مرتبط با CRISPR) به خوبی استفاده می شدند؛ اغلب به اندازه کافی تایید نشده است، از این رو این مقدمه ای در مورد تاریخچه کوتاه مدت اما قوی ویرایش ژنوم در پستانداران است.
مکانیسمی که از طریق آن انواع مختلفی از نوکلئازها اختلال در ژن یا HR را در حضور مولکول های DNA اهدا کننده مناسب افزایش می دهند ، یکسان است. یعنی همه آنها به طور موثری DSB ها را در مکان های ژنومی خاص القا می کنند. پس از آن، مکانیسم های ترمیم سلولی اندوژن، در غیاب (اتصال انتهاهای غیر همولوگ، NHEJ) و یا حضور (ترمیم همولوگوس، HDR) مولکول های DNA دهنده مناسب، با توالی همولوگوس اطراف DSB، این شکاف در ژنوم را پر می کند. بسته به مسیر ترمیم بکار گرفته شده توسط سلول، DSB اصلی می تواند منجر به وقوع اختلال در ژن شود ، که با درج یا حذف نوکلئوتید ها (INDEL ها) از طریق NHEJ یا در رویدادهای ویرایش ژن مرتبط با HDR ، همراه است (شکل 1). در واقع، مزایای قابل اندازه گیری این نوکلئازها در ارتباط با افزایش فرکانس وقایع HR ای که آنها می توانند القا کنند، در مقایسه با تلاش های قبلی بدون استفاده از نوکلئاز، حداقل 1000 برابر (Bibikova و همکاران 2001)تخمین زده می شود (Smithies et al.، 1985 ؛ Brinster و همکاران، 1989).
Zinc‑finger nucleases (ZFN)با وجود اینکه مگانوکلئازهای مخمر اولین مورد برای اصلاح ژنوم پستانداران بودند (Choulika et al.، 1995؛ Cohen-Tannoudji et al.، 1998)، یک تغییر اساسی در متدولوژی در سال 2009 برای چگونگی تولید حیوانات ترانس ژن رخ داد. در یک مطالعه پیشگام، گوترز، جاموب و بولوو و همکارانش از نوکلئازهای انگشت روی (ZFN) برای تولید اولین موش های knockout در دنیا استفاده کردند (Geurts et al.، 2009). رویکرد روش شناختی بر اساس استفاده از دومین اندونوکلئاز DNA از آنزیم محدود کننده باکتری FokI، مهندسی شده با دومین های finger zinc با ظرفیت اتصال پذیری مشخص DNA است که برای هدف گذاری و برش یک محل خاص ژنوم مورد استفاده قرار گرفت. در واقع نوکلئازهای انگشت روی (ZFN) چندین سال پیش طراحی شده بودند ، زمانی که اولین پروتئین کایمریک حاصل از فیوژن دومین اندونوکلئاز FokI با دومین DNA-binding zinc-finger مهندسی شده ، نشان داده شد که به طور موثری برش و HR را در توالی های ژنومی خاص در جنین های زنوپوس القا می کند. (Bibikova et al 2001). تکنولوژی چند سال بعد از این بهبود یافت و نشان داده شد که ZFN مهندسی شده می تواند جهش SCID انسان در سلول ها را از طریق HR توسط یک DNA دهنده خارج کروموزومی با راندمان بالا (بیش از 18 درصد از سلول ها) بدون انتخاب اصلاح کند (Urnov et al 2005).
تولید DSB مورد انتظار، نیازمند دیمریزاسیون دمین اندونوکلئاز FokI است، که نشان می دهد که اثر ترکیبی دو ZFN برای برش موثر DNA در هر دو رشته مورد نیاز است (Mani et al 2005). به زودی روش هایی برای طراحی ZFN برای ویرایش ژنوم ،ژن های پستانداران ایجاد شد (Porteus 2008).
Abstract
Genome editing is now a routine procedure in many mammalian genetics laboratories. The ostensibly short but intense history of genome-editing approaches illustrates how a disruptive technology can universally colonize a field when this new methodology, conceived to alter mammalian genomes at specific locations, is found to efficiently and robustly deliver results. This review summarizes the early development of genome editing using nucleases, from the pioneering experiments using yeast meganucleases, to the latest prokaryotic nucleases used for precise genome manipulation. Gene-editing nucleases belong to one of three known categories:
zinc-finger nucleases (ZFN), transcription activator-like effector nucleases (TALEN), and clustered regularly interspaced short palindromic repeats (CRISPR) and their associated proteins (Cas) tools. All operate on the same principle; they are all capable of inducing a double-strand break (DSB) at a defined genomic sequence that is subsequently corrected by endogenous DNA repair mechanisms. DSBs can be repaired through non-homologous end joining (NHEJ), resulting in small insertions and/or deletions (INDELs) and, hence, often leading to gene disruption. Alternatively, DSBs can be repaired through homology-driven repair (HDR), in the presence of donor homologous DNA sequences, resulting in gene-editing events.
Introduction
The exchange of endogenous genomic sequences for exogenous donor DNA molecules via homologous recombination (HR) is a process that has been known for decades. Pioneering studies by the late Oliver Smithies illustrated how homologous DNA molecules could recombine and correctly insert at defined mammalian chromosomal locations (Smithies et al. 1984, 1985). These findings were instrumental for envisaging and developing gene targeting methodologies in mouse embryonic stem (ES) cells, for which Smithies, together with Mario Capecchi and Martin Evans, were jointly awarded the Nobel Prize in the Physiology or Medicine in 2007 (Mak 2007). In these first experiments, the recorded efficiency of targeted integration by HR in cultured somatic cell lines was relatively low (at best 1 in 1000), even in the presence of selection (Smithies et al. 1985). Nonetheless, the method was at least ten times more efficient than attempting to insert a genomic DNA fragment into its corresponding chromosomal location by HR through direct pronuclear microinjection of mouse fertilized eggs, as demonstrated by the perseverance of Ralph Brinster and collaborators (Brinster et al. 1989).
It became quickly obvious that opening the donor DNA molecule through a double-strand break (DSB) within the region of homology considerably enhanced the frequency of HR within the gene (Kucherlapati et al. 1984; Smithies et al. 1985). A similar observation was confirmed some years later in mouse ES cells when Allan Bradley reported increased targeting efficiencies using insertion versus replacement vectors (Hasty et al. 1991). Therefore, the benefits for the integration efficiency somehow correlated with the promotion of DSBs in the donor DNA template, so exposing homologous sequences at the end of these DNA molecules, a concept that was solidly established. This observation would play a fundamental role in subsequent developments leading to the first genome-editing strategies.
Indeed, the next step towards the establishment of the first genome-editing strategies was based on using I-SceI yeast meganuclease, a rare-cutter endonuclease with a recognition site of 18 base pairs responsible for intron homing in yeast mitochondria (Jacquier and Dujon 1985). 10 years after its discovery, it was shown that I-SceI meganuclease could be used to promote HR in mammalian chromosomes with a frequency two orders of magnitude higher than spontaneous HR (Choulika et al. 1995). The authors demonstrated the efficient repair of I-SceI-induced specific DSBs with donor DNA molecules carrying regions of homology flanking the endogenous I-SceI site in mouse cells, which was probably one of the first attempts of applying genome-editing strategies based on I-SceI meganuclease to produce a DSB for repair by endogenous cellular mechanisms. Subsequently, the introduction of I-SceI restriction sites at a mouse genomic locus by HR in ES cells, and the succeeding expression of this meganuclease in the presence of a replacement vector with homologous DNA sequences surrounding the introduced I-SceI site, resulted in a significant (100-fold stimulation) increase in targeting efficiency (Cohen-Tannoudji et al. 1998). Of course, the procedure was lengthy and cumbersome, requiring two consecutive HR steps and two selection markers. It was also not very practical since the I-SceI-based integration promotion was strictly dependent on the prior introduction of I-SceI recognition sites at the locus to be modified. Moreover, recent reports have suggested that HR by meganuclease-induced DSBs may be locus dependent in mammalian cells (Fenina et al. 2012). Introducing I-SceI meganuclease sites at either end of transgenes and co-microinjecting with the meganuclease also resulted in increased efficiency of transgenesis in medaka fish embryos (Thermes et al. 2002) and Xenopus (Pan et al. 2006), but not in mammalian embryos. Recently, this limitation has been overcome using a modified meganuclease with a nuclear localization signal (NLS), which efficiently mediates germline transgenesis in mouse and porcine embryos (Wang et al. 2014).
The evident limitation of target sequences associated with I-SceI would ultimately be overcome by producing engineered meganucleases with customized recognition sites, once they became commercially available. One of these engineered meganucleases, derived from I-CreI, was used to induce targeted recombination at high frequency at the SCID gene in human cells (Grizot et al. 2009). More recently, another engineered I-CreI, cleaving a 22-bp sequence of the Rag1 gene, was applied to demonstrate effective targeting events in rat and mouse embryos (Menoret et al. 2013).
The recognition that meganucleases were the first to be used for genome-editing purposes, well before other more celebrated and recent genome-editing nucleases (zincfinger nucleases, ZFN; transcription activator-like effector nucleases, TALEN; CRISPR-associated proteins, Cas), is often not sufficiently acknowledged; hence these introductory words in the short but intense history of genome editing in mammals.
The mechanism by which the different types of nucleases enhance gene disruption or HR, in the presence of suitable donor DNA molecules, is the same; namely, they all efficiently promote DSBs at specific genomic locations. Subsequently, the endogenous cellular repair mechanisms, in the absence (non-homologous end joining, NHEJ) or presence (homology-driven repair, HDR) of adequate donor DNA molecules, with homologous sequences surrounding the DSB, will seal this breach in the genome. Depending on the repair route taken by the cell, the original DSB can result in gene disruption events, which are associated with the insertion or deletion of nucleotides (INDELs) through NHEJ, or in gene-editing events, which are associated with HDR (Fig. 1). Indeed, the quantifiable benefits of these nucleases regarding the increase in the frequency of HR events they can promote are estimated to be at least 1000-fold (Bibikova et al. 2001), as compared to previous attempts without nucleases (Smithies et al. 1985; Brinster et al. 1989).
Zinc‑finger nucleases (ZFN) Even though yeast meganucleases were the first to be utilized to edit mammalian genomes (Choulika et al. 1995; Cohen-Tannoudji et al. 1998), a fundamental change in methodology occurred in 2009 that would transform how transgenic animals are produced. In a pioneering study, Guerts, Jacob and Buelow, and colleagues used zinc-finger nucleases (ZFN) to produce the world´s first knockout rats (Geurts et al. 2009). The methodological approach was based on the use of a DNA endonuclease domain from the bacterial restriction enzyme FokI, engineered with zincfinger domains with known DNA-binding capacity, which was used to target and cleave a specific genome location. Zinc-finger nucleases (ZFN) had actually been devised some years earlier when the first chimeric proteins resulting from fusing the FokI endonuclease domain with engineered DNA-binding zinc-finger domains were shown to efficiently cleave and promote HR events at specific genomic sequences in Xenopus embryos (Bibikova et al. 2001). The technology was improved a few years after this and the engineered ZFN were shown to drive the correction of the human SCID mutation in cells by HR with an extrachromosomal DNA donor at high efficiencies (more than 18% of cells) without selection (Urnov et al. 2005).
Generating the expected DSB required dimerization of the FokI endonuclease domain, indicating that the combined effect of two ZFN was required for efficient DNA cleavage at both strands (Mani et al. 2005). Soon, approaches were developed to design ZFN for the genome editing of mammalian genomes (Porteus 2008).
چکیده
مقدمه
Zinc‑finger nucleases (ZFN)
افکتور نوکلئاز شبه فعال کننده رونویسی (TALEN)
تناوبهایِ کوتاهِ پالیندرومِ فاصلهدارِ منظمِ خوشهای( کریسپر) و پروتئین های مرتبط با کریسپر (Cas)
آینده نوکلئازهای ویرایش ژنوم
Abstract
Introduction
Zinc‑finger nucleases (ZFN)
Transcription activator‑like effector nucleases(TALEN)
Clustered regularly interspaced short palindromic repeats (CRISPR) and CRISPR‑associated proteins (Cas)
The future of genome‑editing nucleases
References
