
دانلود رایگان مقاله بدست آوردن چارچوب تعیین منابع برای محاسبات ژنومیک
چکیده
در سال های اخیر محاسبات علمی با استفاده گسترده از محاسبات ابری به دلیل انعطاف پذیری آن در مدیریت منابع محاسباتی تکامل یافته است. در این مقاله، ما روی پردازش داده های ژنومی که به سرعت در حال پیشرفت در تحقیق و فعالیت های پزشکی است، تمرکز می کنیم. از ویژگی های اصلی این مجموعه داده، نه تنها تعداد فایل های ژنوم موجود در حال تبدیل شدن بسیار بزرگ است، بلکه هر یک از مجموعه داده ها به تنهایی به طور قابل توجهی بزرگ بوده، و بیش از ده گیگابایت می باشد. از این رو، پردازش بخش بزرگی از داده های ژنومی مبتنی بر ابر، تاثیر قابل توجهی در منابع شبکه دارند، زیرا هر پردازش نیاز به انتقال دهها گیگابایت به گره های محاسبه دارد. برای بررسی این موضوع، در این مقاله ما یک چارچوب تعیین منابع پیشنهاد می کنیم که عوامل تصمیم گیری با اطلاعات مورد نیاز برای انتخاب مناسب ترین گره های محاسبه را فراهم می کند. ما تابع تعیین منابع را به صورت توزیع شده اجرا کرده، و به طور گسترده آن را در یک بستر آزمایشی آزمایشگاه متشکل از حدود 70 گره تست کردیم. ما دریافتیم اورهد راه حل پیشنهادی در مقایسه با مقدار داده منتقل شده، ناچیز است.
1. مقدمه
در سال های اخیر، اجرای سیستم عامل محاسبات علمی با استفاده از محاسبات مبتنی بر خوشه محلی و کاربرد محاسبات شبکه ای توزیع یافته، تکامل یافته، و آخرین دستاورد، استفاده از زیرساخت های محاسبات ابری می باشد [13]. این تکامل است به دلیل انعطاف پذیری بهبود یافته در مدیریت منابع محاسباتی می باشد که این آخرین پارادایم را نسبت به سایر راهها ارائه می دهد. با این حال، محاسبات علمی با برنامه های کاربردی معمولی مبتنی بر ابر، اعم از میزبانی سرور چند رسانه ای و استقرار تجهیزات ذخیره سازی، بسیار متفاوت است. تفاوت اصلی شامل حجم اطلاعات مدیریت شده توسط برنامه های علمی و یا CPU مورد نیاز، است که نسبت به سایر کاربردها، بسیار بیشتر می باشد. علاوه بر این، برنامه های کاربردی محاسبات علمی بسیار ناهمگن هستند. برای مثال، آنها ممکن است از سیستم عامل های مورد استفاده برای ذخیره و پردازش خروجیهای آزمایشهای فیزیکی دارای انرژی بالا، که نیاز به امکانات تجربی به منظور جمع آوری مقدار زیادی از داده ها به موقع ، یا شبیه سازی تغییرات آب و هوا دارد، استفاده کنند. این کار نیاز به عملکرد محاسباتی بالا، و یا پردازش مجموعه داده های ژنومی دارد، که ممکن است مقادیر بیشتری نسبت به منابع محاسباتی لازم برای اجرای تک، با تعداد بسیار بیشتر اجراها، نیاز داشته باشد که این مسئله سبب می شود که اندازه ورودی فایل ها به راحتی به دهها گیگابایت برسد.
در این مقاله، ما روی پردازش داده های ژنومی کار می کنیم. این موضوع با توجه به کاهش در هزینه های تعیین توالی DNA ، به سرعت در فعالیت های پژوهشی و پزشکی در حال پیشرفت و گسترش است.[1] از ویژگی های اصلی این مجموعه داده، نه تنها تعداد فایل های ژنوم موجود در حال تبدیل شدن بسیار بزرگ است، بلکه هر یک از مجموعه داده ها به تنهایی به طور قابل توجهی بزرگ بوده، و بیش از ده گیگابایت می باشد. به این مساله تحت عنوان Big2 اشاره شده است، و اهمیت آن در طول زمان افزایش می یابد. در واقع، انتظار می رود که در چند سال آینده تمام نوزادان تعیین توالی خواهند شد و علوم پزشکی، بر نتایج پردازش ژنوم پردازش استوار خواهد شد. واضح است که این مساله واقع بینانه نیست که فرض کنیم که هر بیمارستان قادر به دست آوردن یک مرکز محاسبات بزرگ (ابر خصوصی) برای رفع نیاز به تقاضای پردازش داخلی خواهد بود. بنابراین، استفاده از خدمات پردازش عمومی مبتنی بر ابر راه حل واضح خواهد بود[11]. از این رو، بدیهی است که مدیریت مناسب داده های ژنومی نه تنها برای خدمات ذخیره سازی، بلکه همچنین برای پردازش آنها و انتقال آنها ضروری خواهد بود. در واقع، پردازش داده های ژنومی مبتنی بر ابر تاثیر قابل توجهی در منابع شبکه دارند، زیرا هر درخواست پردازش سبب انتقال گیگابایت ها از داده ها به گره های محاسبه در مراکز داده ی ارائه دهندگان ابر خواهد شد.
در این مقاله یک پروتکل تعیین منابع ابر [22] [29]، با ارائه عوامل تصمیم گیری با تعدادی از اطلاعات زمینه پیشنهاد خواهد شد. این اطلاعات ممکن است شامل موقعیت و در دسترس بودن منابع پردازش (CPU، RAM، ذخیره سازی)، داده های ورودی به پردازش شامل فایل های کمکی (به عنوان مثال حاشیه نویسی [12])، و فایل های تصویری از ماشین های مجازی (VMS) میزبانی بسته های نرم افزاری ژنومی مورد استفاده برای پردازش ژنومیک داده، باشد. در واقع، نوع پردازش ژنومی (اجرا از طریق ترکیبی از برنامه های مختلف، مربوط به خط لوله ژنومی) است که می تواند در حین یک ژنوم تک بسیار بزرگ اجرا شود. فقط به یک ایده، خواننده علاقه مند می تواند یک لیست غیر جامع گزارش شده در جدول 1، از [12] را پیدا کند. بنابراین، واقع بینانه نیست که انتظار داشته باشیم هر مرکز داده از یک ارائه دهنده ابر، توانایی میزبانی همه ماشین های مجازی مورد نیاز برای هر درخواست ممکن را به طور همزمان داشته باشد.
پروتکل تعیین ما از توابع ارائه شده توسط گام های بعدی در چارچوب انتشار سیگنال (NSIS) استفاده می کند [2] [3]. پس ما ابتدا با استفاده از توابع ارائه شده توسط گسترش offpath که به تازگی تعریف شده، مجموعه پروتکلهای NSIS اجرا شده را ارائه [8] و در ادامه برای آن تعریف بهتری ارائه خواهیم داد[9]. این راه حل اجازه می دهد تا انتشار سیگنال را در مناطق شبکه ای به شکل دلخواهش نزدیک شود. با اعمال قابلیت رهگیری از لایه انتقال سیگنالینگ NSIS، این پروتکل انتشار سیگنالینگ، بسیار کارآمد خواهد شد و ممکن است اجازه ی پیدا کردن منابع نزدیک به یک مسیر شبکه داده را بدهد. در سناریوی در نظر گرفته، مسیر تحت بررسی می تواند، به عنوان مثال، یکی از اتصالات مخزن ذخیره سازی تصاویر VM مورد نیاز و مرکز داده های ذخیره سازی داده های ورودی باشد. از آنجایی که خوشه های محاسباتی در مراکز داده قادر به میزبانی ماشینهای مجازی هستند لذا نمی توانند در مسیر IP اتصال دو سرور (فقط روتر در مسیر قرار دارد) قرار گیرند،پس از یک پروتکل سیگنالینگ با قابلیت تعیین مسیر برای تعیین مراکز داده با هر دو قابلیت محاسبات کافی و موقعیت مناسب به منظور به حداقل رساندن ترافیک شبکه استفاده می شود. هنگامی که این اطلاعات به مرکز تصمیم فرستاده می شود، می تواند یک الگوریتم بهینه سازی برای انتخاب مرکز داده را اجرا کند.
راه حل ما مي تواند بعنوان راه تشخيص منابع حل نقطه به نقطه با قابليت كوپله شدن با آدرس هاي شبكه مشخص بر پايه ان اس اي اس تقسيم بندي شود. همانگونه كه در ٢٦ بيان شد روش هاي ملاسيك نقطه به نقطه قابليت ارائه راه حل هاي بر پايه نزديكي ندارند و تنها سعي در محدود كردن اورهد شبكه دارتد. روش هاي شاخه اي تشخيص مناسب منابع مربوط به يك سوپر نقطه را ممكن مي كنند اما وقتي بيش از دو سوپر نقطه در ميان باشند مشكل به اين سادگي نخواهد بود. راه حل هاي ديگري كه اخيرا ارائه شده اند از اونتولوژي مناسب براي تشخيص سرويس استفاده مي کنند. بدون اينكه به مشكلات نزديكي مسير توجه كنند. در ٢٤ نويسندگان يك لايه خلاصه كننده را جهت تشخيص مناسب ترين منابع زيرساخني معرفي نمودند كه بعدا در راه حل برپايه قيود كه در فضاي ابري چند منبعه استفاده مي شود. با اين وجود مشكلات نزديكي به طرز دقيقي رسيدگي نميشوند و تنها با ارضاي نيازهاي مكاني رسيدگي ميشوند. راه حل هاي ديگري نيز به طور خاص براي پلتفرم هاي محاسبه شبكه اي مثل ٢١ ٢٧ طراحي شده اند. در ٢١ نيز تلاش به جهت به حداقل رساندن همپوشاني اطلاعات شبكه است و نه براي پيدا كردن گره هاي نزديك به مكان ها يا مسيرهاي خاص.
ما راه حل تعیین ارائه شده در یک بستر آزمایشی آزمایشگاه متشکل از حدود 70 گره را اجرا کردیم، و آزمایشات گسترده ای در این باره انجام دادیم. نتایج به دست آمده ثابت کرد که مقدار اورهد شبکه در راه حل پیشنهادی در مقایسه با اندازه فایل های داده رد و بدل شده ناچیز است.
مقاله بصورت زیر مرتب شده است. بخش دوم برخی از پیش زمینه های NSIS، و سناریوی مرجع ارائه شده توسط پروژه ARES [10] را نشان می دهد. بخش سوم اطلاعات الگوریتمی و پروتکل راه حل ما را فراهم می کند ، و در بخش چهارم نتایج آزمایشات آزمایشگاه ارائه شده است. در نهایت، در بخش پنجم ملاحظات و توضیحات ما ارائه شده است.
2. پیش زمینه و سناریوی مرجع
A. پیش زمینه پروتکل های NSIS و اجرای off-path
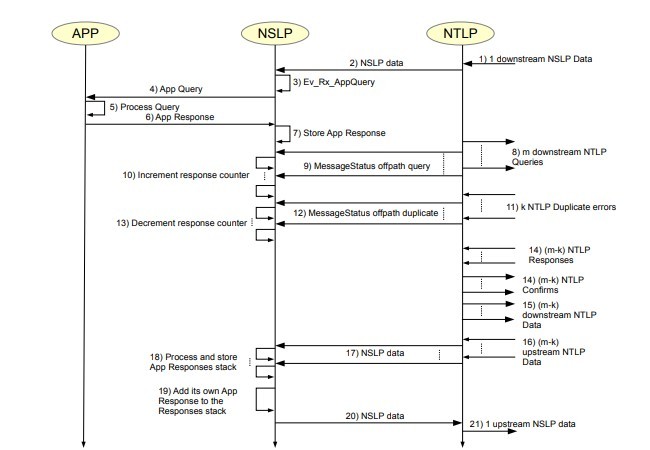
مجموعه پروتکل NSIS ، توابع سیگنالینگ را به دو لایه تقسیم می کند [3]. لایه فوقانی، به نام پروتکل لایه ی سیگنالینگ NSIS (NSLP)، وظیفه ی پیاده سازی منطق برنامه سیگنالینگ را بر عهده دارد. لایه پایین تر، به نام پروتکل لایه انتقال NSIS (NTLP)، وظیفه ی ارائه پیام NSLP به گره NSIS بعدی در مسیر به سوی هدف مشخص را دارد. سیگنالینگ انتقال اینترنت عمومی (GIST) نسخه IETF تعریف شده ی [5] NTLP است. خدمات انتقال GIST تنها با استفاده از پروتکل های موجود در TCP / IP مقدور می باشد. GIST تنها پیام های NSLP نقطه به نقطه بین جفت همسایه های گره سیگنالینگ NTLP را می رساند، در حالی که توابع سیگنالینگ انتها به انتها، در صورت نیاز، توسط NSLP ارائه شده است.
Abstract
In recent years scientific computing has evolved into a massive usage of cloud computing, due to its flexibility in managing computing resources. In this paper, we focus on genomic data processing, which is rapidly gaining momentum in research and medical activities. The main characteristics of these data sets is that not only the number of available genome files is becoming extremely large, but also each individual data set is significantly large, in the order of tens of GB. Hence, a wide diffusion of cloud-based genomic data processing will have a significant impact on network resources, since each processing request will require the transfer of tens of GBs into computing nodes. To face this issue, in this paper we propose a resource discovery framework which provides decision agents with the needed information for selecting the most suitable computing nodes. We have implemented this resource discovery function in a distributed fashion, and extensively tested it in a lab testbed consisting of about 70 nodes. We found that the overhead of the proposed solution is negligible in comparison with the amount of transferred data.
I. INTRODUCTION
In recent years, the implementation of scientific computing platforms have evolved from the use local cluster-based computing, to the use of distributed grid computing and, more recently, of cloud computing infrastructure [13]. This evolution is due to the improved flexibility in managing computing resources that this latest paradigm offers over the other ones. However, scientific computing is very different from typical cloud-based applications, ranging from hosting multimedia servers to deploying storage facilities. The main differences include the volume of data managed by scientific applications and/or CPU requirements, which could be of some orders of magnitude larger than in other types of applications. In addition, scientific computing applications are highly heterogeneous. For example, they may consist of platforms used to storage and process the output of high energy physics experiments, which need to be co-located with experimental facilities in order to timely collect the huge amount of data, or climate changes simulations, which needs high performance computing architectures, or genomic data sets processing, which may require more modest amounts of computing resources for a single execution, with a highly increasing number of executions to be implemented, with a size of the input files easily reaching tens of GBs.
In this paper, we focus on genomic data processing, which is rapidly gain momentum in research and medical activities, due to the reduction in the DNA sequencing costs [1]. The main characteristics of these data sets is that not only the number of available genome files is becoming extremely large, but also each data set is significantly large, in the order of tens of GB. This problem is referred to as Big2 data problem, and its importance increases over time. In fact, it is expected that in the next few years all newborns will be sequenced and the medical science will build upon genome-processing outcomes. Clearly, it is not realistic to assume that each hospital will be able to acquire a large computing facility (private cloud) to cope with the internal processing demand. Thus, the use of public cloud-based processing services will be the obvious solution [11]. Hence, it is evident that the suitable management of genomic data will be essential not only for storage services, but also for their processing and their transfer. In fact, a wide diffusion of cloud-based genomic data processing will have a significant impact on network resources, since each processing request will require the transfer of many GBs of data into computing nodes in data centers of cloud providers.
In this paper we propose a discovery protocol of cloud resources [22][29], by providing decision agents with a number of context information. This information may consist of position and availability of processing resources (CPU, RAM, storage), input data to be processed, auxiliary files (e.g. annotations [12]), and image files of the virtual machines (VMs) hosting the genomic software packages used to process genomics data. In fact, the types of genomic processing (implemented through the combination of different programs, referred to as genomic pipelines) that can be executed over a single genome is quite large; just to have an idea, the interested reader can find a non-exhaustive list reported in Table 1 of [12]. Thus, it is not realistic to expect that each datacenter of a cloud provider can simultaneously host all VMs needed for any possible request.
Our discovery protocol leverages the functions offered by the Next Steps in Signaling (NSIS) framework [2][3]. In more detail, we use the functions offered by the recently defined offpath extension to the NSIS protocol suite, initially presented in [8] and further refined in [9]. This solution allows disseminating signaling over network areas of nearly arbitrary shape. By leveraging the interception capabilities of the signaling transport layer of NSIS, this signaling dissemination protocol is highly efficient, and may allow finding resources which are close to a given network path. In the considered scenario, the path under consideration could be, for instance, the one connecting the repository storing the needed VM images and the data center storing the input data. Since computing clusters in datacenters able to host VMs cannot be on the IP path connecting two servers (only routers are located on path), a signaling protocol with off-path discovery capabilities is used to discover data centers with both sufficient computing capabilities and a position suitable in order to minimize the overall network traffic exchanged. When this information is reported back to a decision agent, the latter can execute an optimization algorithm for selecting a datacenter.
Our solution can be classified as a peer-to-peer (P2P) resource discovery solution [25], with the feature that, being based on NSIS, it is able to couple with specific network paths. Classic P2P-based approaches [28][23] offer little or no support to provide proximity-based solutions, as stated in [26], trying just to limit the network overhead. Hierarchical solutions enable the efficient discovery of the resources belonging to one super-peer, but when more than one super-peers are involved, the problem is not trivial. Other solutions, recently proposed, make use of a proper ontology for service discovery [18][19][20], without considering path proximity issues. In [24], the authors propose an abstraction layer to discover the most appropriate infrastructure resources, which is then used in constraint-based approach applied to a multi-provider cloud environment. However, proximity issues are loosely handled, by simply referring to location requirements. Other solutions have been specifically designed for grid computing platforms, such as [21][27]. In [21], routing hops are considered, but they are relevant to the grid overlay. In [27], again the effort is to minimize the amount of message on the grid overlay, and not to find nodes close to specific locations or paths.
We have implemented the proposed discovery solution in a lab testbed consisting of about 70 nodes, and have executed extensive tests. The obtained results prove that the network overhead of the proposed solution is negligible when compared with the size of data files to be exchanged.
The paper is organized as follows. Section II presents some background on NSIS, and the reference scenario, which is represented by the project ARES [10]. Section III provides algorithmic and protocols details of our solution, and Section IV presents the results of lab experiments. Finally, we draw our concluding remarks in Section V.
II. BACKGROUND AND REFERENCE SCENARIO
A. Background on NSIS protocols and off-path extension
The NSIS protocol suite divides the signaling functions into two layers [3]. The upper layer, called NSIS Signaling Layer Protocol (NSLP), implements the application signaling logic.
چکیده
1. مقدمه
2. پیش زمینه و سناریوی مرجع
A. پیش زمینه پروتکل های NSIS و اجرای off-path
B. سناریوی مرجع: پروژه ی ARES
3. تعیین چارچوب
4. تخمین عملکرد
5. نتیجه گیری
منابع
Abstract
1. INTRODUCTION
2. BACKGROUND AND REFERENCE SCENARIO
A. Background on NSIS protocols and off-path extension
B. Reference scenario: the ARES project
3. DISCOVERY FRAMEWORK
4. PERFORMANCE EVALUATION
5. CONCLUSION
REFERENCES
