
دانلود رایگان مقاله بررسی قطعه بندی تصویر تومور مغزی مبتنی بر MRI با استفاده از روش های یادگیری
چکیده
قطعه بندی تومور مغزی یکی از کارهای مهم در زمینه ی پردازش تصویر پزشکی است. تشخیص زودهنگام تومورهای مغزی دراحتمال بهبود با درمان و افزایش نرخ زنده ماندن بیماران نقش مهمی ایفا می کند. قطع بندی تومورهای مغزی برای تشخیص سرطان به صورت دستی (توسط انسان)، از میان تعداد زیادی از تصاویر MRI تولید شده در روتین های پزشکی کاری دشوار و وقتگیر است. یک نیاز اساسی برای قطعه بندی تصویر تومور مغزی به صورت خودکار وجود دارد. هدف از این مقاله، ارائه ی یک بررسی بر روی روش های قطعه بندی تصویر تومور مغزی مبتنی بر MRI است. به تازگی، استفاده از روشهای یادگیری عمیق برای قطعه بندی خودکار محبوبیت پیدا کرده اند، چرا که این روش ها به نتایج پیشرفته و جدیدی دست می یابند و می توانند بهتر از روش های دیگر به این مسئله رسیدگی کنند. روش های یادگیری عمیق همچنین می توانند پردازش کارآمد را فعال کنند و ارزیابی قابل مشاهده و هدفمندی از حجم عظیمی از داده های تصویری مبتنی بر MRI را ممکن سازند. تعدادی از مقالات بررسی (review) وجود دارند که بر روش های قدیمی(سنتی) برای قطعه بندی تصویر تومور مغزی مبتنی بر MRI تمرکز کرده اند. ولی برخلاف دیگران، ما در این مقاله بر روی روند اخیر روش های یادگیری عمیق در این زمینه تمرکز می کنیم. ابتدا، معرفی تومورهای مغزی و روش هایی برای قطعه بندی تومور مغزی داده می شوند. سپس، الگوریتم های جدید با تمرکز بر روی روند اخیر روش های یادگیری عمیق مورد بررسی قرار می گیرند. در نهایت، یک ارزیابی از وضعیت فعلی ارائه شده و پیشرفتهای آینده جهت استانداردسازی روش های قطعه بندی تومور مغزی مبتنی بر MRI در روتین های روزانه ی پزشکی در نظر گرفته شوند.
1. مقدمه
سرطان را می توان به عنوان رشد و تقسیم غیر قابل کنترل و غیرطبیعی سلول ها در بدن تعریف کرد. وقوع این رویداد، یعنی رشد و تقسیم غیرطبیعی سلول ها در بافت مغزبه صورت یک توده، تومور مغزی نامیده می شود. در حالی که تومورهای مغزی خیلی رایج نیستند، ولی یکی از انواع بسیار کُشنده ی سرطان محسوب می شوند.
بسته به منشأ اولیه ی آنها، تومورهای مغزی می توانند به عنوان تومورهای مغزی اولیه یا تومورهای مغزی متاستاتیک )ثانویه) در نظر گرفته شوند. در مورد اول (تومورهای مغزی اولیه) منشأ سلول ها، سلولهای بافت مغزی هستند که در موارد متاستاتیک، سلول ها در قسمت دیگری از بدن سرطانی می شوند و تا مغز گسترش می یابند. گلیوم ها انواعی از تومورهای مغزی هستند که از سلول های گلیال ایجاد می شوند. آنها نوع اصلی تومورهای مغزی هستند که در حال حاضر پژوهش های قطعه بندی تومور مغزی بر روی آن تمرکز دارند. اصطلاح گلیوم یک اصطلاح کلی است که برای توصیف انواع مختلفی از گلیوما ها اعم ازگلیومای درجه پایین مانند آستروسیتوم ها و الیگودندروگلیوما به درجه بالا (درجه یIV ) گلیوبالستومای چند شکلی (GBM) که تهاجمی ترین و رایج ترین تومور مغزی بدخیم اولیه است. جراحی، شیمی درمانی و پرتو درمانی روش های استفاده شده ، معمولا به صورت ترکیبی برای درمان گلیوما هستند.
تشخیص زودهنگام گلیوما نقش مهمی را در احتمال بهبود از طریق درمان ایفا می کند. روش های تصویربرداری پزشکی از قبیل توموگرافی (CT)، توموگرافی رایانه ای با گسیل تک فوتون (SPECT) ، توموگرافی با گسیل پوزیترون (PET) ، طبف سنجی رزونانس مغناطیسی (MRS) و طیف سنجی تشدید مغناطیسی (MRI) همه برای ارائه ی اطلاعات ارزشمندی در مورد شکل، اندازه، مکان و متابولسیم تومورهای مغزی برای کمک به تشخیص مورد استفاده قرار می گیرند. در حالی که این روش ها به صورت ترکیبی مورد استفاده قرار می گیرند تا اطلاعات دقیق ترین در مورد تومورهای مغزی آماده کنند، بهعلت کنتراست خوب از بافت نرم و همچنین دسترسی پذیری گسترده یMRI ، به عنوان روش استاندارد در نظر گرفته شده است.MRI یک روش تصویربرداری غیر-تهاجمی از موجودات زنده در داخل بدن است که از سیگنال های فرکانس رادیویی برای تحریک بافت های هدف استفاده می کند تا تصاویر داخلی آنها را تحت تاثیر یک میدان مغناطیسی بسیار قوی تولید نماید. تصاویری از دنباله های مختلف MRI به وسیله ی تغییر میزان تحریک و دفعات تکرار در حین گرفتن تصویر، تولید می شوند. این حالت های مختلف MRI ، انواع مختلف تصاویر کنتراست بافت را تولید می کنند، بدین ترتیب اطلاعات ارزشمند و با ساختاری را فراهم نموده و تشخیص و قطعه بندی تومورها را در زیر مناطق خود ممکن می سازد. چهار مدل استاندارد MRI برای تشخیص گلیوما مورد استفاده قرار می گیرند که عبارتند از: MRI با وزن دهی T1( T1)،MRI با وزن دهی T2 (T2)، MRI با وزن دهی T1 با ارتقای کنتراست گادولینیم (Gd-T1) وجبران معکوس مایع رقیق شده) (FLAIR )شکل 1 را ببینید(. در حین گرفتن MRI ،حدود صد و پنجاه قطعه تصویر 2-بُعدی تولید م یشوند تا یک توده ی 3-بُعدی از مغز نمایش داده شود، اگرچه این تعداد تصاویر می تواند از یک دستگاه به دستگاه دیگر متفاوت باشد. علاوه بر این، هنگامی که قطعه ها از مدل های استاندارد مورد نیاز ترکیب می شوند برای تشخیص داده ها بسیار شلوغ و پیچیده می گردند.
به طور کلی، تصاویر T1 برای تشخیص بافت های سالم مورد استفاده قرار می گیرند، در حالی که تصاویر T2 برای تعیین منطقه ی ورم که سیگنال روشنی روی تصویر تولید می کند، مورد استفاده قرار می گیرند. در تصاویر Gd-T1 ،محدوده ی مرزی تومور می تواند به سادگی به وسیله ی سیگنال روشن ایجاد شده توسط عامل کنتراست انباشته شده (یون های گادولینیم) در منطقه سلولی فعال بافت تومورتشخیص داده شود. از آنجایی که سلول های نکروتیک با عامل کنتراست در تعامل نیستند، آنها می توانند به وسیله ی بخش های فوق متراکم و شدید ماده ی محرک در هسته ی تومور مشاهده شوند که این امر باعث می شود تا جدا کردن و قطعه بندی آنها از منطقه ی سلول های فعالی که در همان دنباله هستند، به سادگی ممکن شود. در تصاویر FLAIR ،سیگنال مولکول های آب تحت فشار قرار می گیرد که به تشخیص منقطه ی تورم از مایع مغزی نخاعی (CSF) کمک کند.
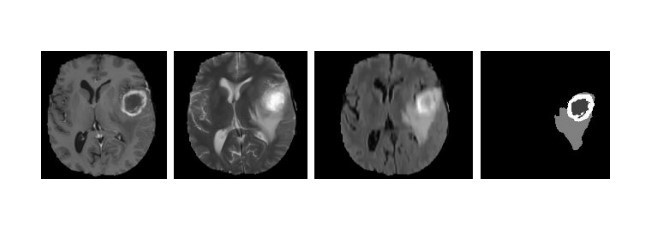
قبل از اعمال هر گونه درمان، قطعه بندی تومور حیاتی است تا از بافت های سالم در حین درمان در حالی که سلولهای تومور نیز تخریب و نابود می شوند، محافظت کند. قطعه بندی تومور شامل تشخیص، تعیین دقیق و جداسازی بافت های تومور، از قبیل سلول های فعال، هسته ی نکروتیک و تورم (شکل 2) از بافت های عادی مغز است که شامل ماده ی خاکستری (GM) ، ماده ی سفید (WM )و CSF می باشد. در روال بالینی فعلی، این کارشامل تفسیر دستی (توسط انسان) و قطعه بندی تعداد زیادی از تصاویر MRI چند حالتی است. با این حال، از آنجایی که قطعه بندی به صورت دستی، یک پروسه بسیار وقتگیر است، از این رو توسعه ی روش های قطعه بندی قوی به صورت خودکار، برای فراهم نمودن یک قطعه بندی کارآمد و هدفمند، در سال های اخیر به یک حوزه ی تحقیقاتی جالب و محبوب تبدیل شده است. امروزه عملکرد بالا در قطعه بندی با استفاده از روشهای یادگیری عمیق به دست آمده، که باعث می شود آنها گزینه ی خوبی برای دستیابی به این هدف تبدیل گردند.
ادامه ی این مقاله به صورت زیر سازماندهی شده است: ما در ابتدا به طور خلاصه روش های قطعه بندی تصویر تومور مغزی را در بخش 2 مرور می کنیم. سپس، در بخش 3 ، به ویژه، بر روی روش های مبتنی بر الگوریتم های یادگیری عمیق تمرکز می کنیم، که نتایج جدید روشهای ارائه شده در سالهای اخیر را آماده می کند. به طور خاص، طرح های مختلف روشهای یادگیری عمیق و عملکرد آنها را با هم مقایسه می کنیم. در نهایت، به عنوان نتیجه گیری، ما وضعیت فعلی این حوزه را برآورد نموده و مسیرهای آینده را برای توسعه آماده می کنیم.
2. روش های قطعه بندی تصویری تومور مغزی
روش های قطعه بندی تصویر تومور مغزی می توانند به صورت روش های دستی، روش های نیمه خودکار و روش های تمام خودکاربر اساس سطح تعامل مورد نیاز با کاربردسته بندی می شوند.
2.1. روش های قطعه بندی دستی
قطعه بندی دستی نیاز دارد تا رادیولوژیست از اطلاعات چند وجهی ارائه شده توسط تصاویر MRI به همراه دانش کالبد شناسی و فیزیولوؤژیکی خود استفاده کند که از طریق آموزش و تجربه به دست آورده است. این رویه، رادیولوژیست را درگیر می کند که قسمت های چند گانه تصاویررا قسمت به قسمت بررسی کند، تا تومور را تشخیص داده و با دقت به دورمناطق تومور به صورت دستی خط بکشد (ترسیم کند). صرفنظر از اینکه این کار بسیار وقتگیر است، قطعه بندی به صورت دستی به رادیولوژیست نیز وابسته است و نتایج قطعه بندی به متغیرهای ارزیاب بزرگ درونی و بیرونی بستگی دارد. با این حال، قطعه بندی دستی به طور گسترده ای برای ارزیابی نتایج روش های نیمه-خودکار و تمام خودکار مورد استفاده قرار می گیرند.
2.2. روش های قطعه بندی نیمه-خودکار
روش های قطعه بندی نیمه-خودکار برای سه هدف اصلی نیاز به تعامل با کاربر دارند: راه اندازی، مداخله یا پاسخ بازخورد و ارزیابی. راه اندازی به طور کلی به وسیله ی تعریف یک منطقه ی مورد علاقه (ROI) یا مورد نظرانجام شده است، شامل منطقه تومور تقریبی برای یک الگوریتم خود جهت پردازش است. پارامترهای روش های پیش پردازش همچنین می توانند متناسب با تصاویر ورودی تنظیم شوند. علاوه بر راه اندازی ، الگوریتم های خودکار می توانند طی فرایند با دریافت بازخورد و فراهم کردن تنظیمات در پاسخ، برای کسب نتیجه مطلوب هدایت شود.علاوه بر این، کاربر می تواند نتایج را ارزیابی کند و در صورت عدم رضایت ، روند را اصلاح یا تکرار کند.
Abstract
Brain tumor segmentation is an important task in medical image processing. Early diagnosis of brain tumors plays an important role in improving treatment possibilities and increases the survival rate of the patients. Manual segmentation of the brain tumors for cancer diagnosis, from large amount of MRI images generated in clinical routine, is a difficult and time consuming task. There is a need for automatic brain tumor image segmentation. The purpose of this paper is to provide a review of MRI-based brain tumor segmentation methods. Recently, automatic segmentation using deep learning methods proved popular since these methods achieve the state-of-the-art results and can address this problem better than other methods. Deep learning methods can also enable efficient processing and objective evaluation of the large amounts of MRI-based image data. There are number of existing review papers, focusing on traditional methods for MRI-based brain tumor image segmentation. Different than others, in this paper, we focus on the recent trend of deep learning methods in this field. First, an introduction to brain tumors and methods for brain tumor segmentation is given. Then, the state-of-the-art algorithms with a focus on recent trend of deep learning methods are discussed. Finally, an assessment of the current state is presented and future developments to standardize MRI-based brain tumor segmentation methods into daily clinical routine are addressed.
1. Introduction
Cancer can be defined as the uncontrolled, unnatural growth and division of the cells in the body. Occurrence, as a mass, of these unnatural cell growth and division in the brain tissue is called a brain tumor. While brain tumors are not very common, they are one of the most lethal cancers1 .
Depending on their initial origin, brain tumors can be considered as either primary brain tumors or metastatic brain tumors. In primary ones, the origin of the cells are brain tissue cells, where in metastatic ones cells become cancerous at any other part of the body and spread into the brain. Gliomas are type of brain tumors that originate from glial cells. They are the main type of brain tumors that current brain tumor segmentation research focuses on. The term glioma is a general term that is used to describe different types of gliomas ranging from low-grade gliomas like astrocytomas and oligodendrogliomas to the high grade (grade IV) glioblastoma multiform (GBM), which is the most aggressive and the most common primary malignant brain tumor2 . Surgery, chemotherapy and radiotherapy are the techniques used, usually in combination, to treat gliomas3 .
Early diagnosis of gliomas plays an important role in improving treatment possibilities. Medical Imaging techniques such as Computed Tomography (CT), Single-Photon Emission Computed Tomography (SPECT), Positron Emission Tomography (PET), Magnetic Resonance Spectroscopy (MRS) and Magnetic Resonance Imaging (MRI) are all used to provide valuable information about shape, size, location and metabolism of brain tumors assisting in diagnosis. While these modalities are used in combination to provide the highest detailed information about the brain tumors, due to its good soft tissue contrast and widely availability MRI is considered as the standard technique. MRI is a non-invasive in vivo imaging technique that uses radio frequency signals to excite target tissues to produce their internal images under the influence of a very powerful magnetic field. Images of different MRI sequences are generated by altering excitation and repetition times during image acquisition. These different MRI modalities produce different types of tissue contrast images, thus providing valuable structural information and enabling diagnosis and segmentation of tumors along with their subregions4 . Four standard MRI modalities used for glioma diagnosis include T1-weighted MRI (T1), T2-weighted MRI (T2), T1-weighted MRI with gadolinium contrast enhancement (T1-Gd) and Fluid Attenuated Inversion Recovery (FLAIR) (see Fig. 1). During MRI acquisition, although can vary from device to device, around one hundred and fifty slices of 2D images are produced to represent the 3D brain volume. Furthermore, when the slices from the required standard modalities are combined for diagnosis the data becomes very populated and complicated.
Generally, T1 images are used for distinguishing healthy tissues, whereas T2 images are used to delineate the edema region which produces bright signal on the image. In T1-Gd images, the tumor border can easily be distinguished by the bright signal of the accumulated contrast agent (gadolinium ions) in the active cell region of the tumor tissue. Since necrotic cells do not interact with the contrast agent, they can be observed by hypo intense part of the tumor core making it possible to easily segment them from the active cell region on the same sequence. In FLAIR images, signal of water molecules are suppressed which helps in distinguishing edema region from the Cerebrospinal Fluid (CSF).
Before applying any therapy, it is crucial to segment the tumor in order to protect healthy tissues while damaging and destroying tumor cells during the therapy. Brain tumor segmentation involves diagnosing, delineating and separating tumor tissues, such as active cells, necrotic core and edema (Fig. 2) from normal brain tissues including Gray Matter (GM), White Matter (WM) and CSF. In current clinical routine, this task involves manual annotation and segmentation of large amount of multimodal MRI images. However, since manual segmentation is a very time consuming procedure, development of robust automatic segmentation methods, to provide efficient and objective segmentation, became an interesting and popular research area in recent years5 . Current high segmentation performances obtained by deep learning methods make them good candidates for achieving this task.
The rest of the paper is organized as follows: First we briefly review methods for brain tumor image segmentation in section 2. Then, in section 3, we especially focus on methods based on deep learning algorithms, which provide the state-of-the-art results in recent years. In particular, we compare designs of different deep learning methods and their performances. Finally, in conclusions, we assess the current state-of-the-art and provide future directions for development.
2. Methods for Brain Tumor Image Segmentation
Brain tumor segmentation methods can be classified as manual methods, semi-automatic methods and fully automatic methods based on the level of user interaction required6 .
2.1. Manual Segmentation Methods
Manual segmentation requires the radiologist to use the multi-modality information presented by the MRI images along with anatomical and physiological knowledge gained through training and experience. Procedure involves the radiologist going through multiple slices of images slice by slice, diagnosing the tumor and manually drawing the tumor regions carefully. Apart from being a time consuming task, manual segmentation is also radiologist dependent and segmentation results are subject to large intra and inter rater variability7 . However, manual segmentations are widely used to evaluate the results of semi-automatic and fully automatic methods.
2.2. Semi-Automatic Segmentation Methods
Semi-automatic methods require interaction of the user for three main purposes; initialization, intervention or feedback response and evaluation8 . Initialization is generally performed by defining a region of interest (ROI), containing the approximate tumor region, for the automatic algorithm to process. Parameters of pre-processing methods can also be adjusted to suit the input images. In addition to initialization, automated algorithms can be steered towards a desired result during the process by receiving feedbacks and providing adjustments in response. Furthermore, user can evaluate the results and modify or repeat the process if not satisfied.
چکیده
1. مقدمه
2. روش های قطعه بندی تصویری تومور مغزی
2.1 روش های قطعه بندی دستی
2.2 روش های قطعه بندی نیمه-خودکار
2.3 روش قطعه بندی تمام خودکار
2.3.1 چالش ها
2.3.2 مجموعه داده BRATS
2.3.3 انواع روش های قطعه بندی تومور مغزی خودکار
2.3.4 خط لوله های پردازش در روش خودکار
3. روش های یادگیری عمیق
4. نتیجه گیری
منابع
Abstract
1. Introduction
2. Methods for Brain Tumor Image Segmentation
2.1. Manual Segmentation Methods
2.2. Semi-Automatic Segmentation Methods
2.3. Fully Automatic Segmentation Methods
2.3.1. Challenges
2.3.2. BRATS Dataset
2.3.3. Types of Automatic Brain Tumor Segmentation Methods
2.3.4. Processing Pipelines of Automatic Methods
3. Deep Learning Methods
4. Conclusions
References
