
دانلود رایگان مقاله سیستم نمادهای گرافیکی زیست شناسی
نمودارهای مداری و نمودار زبان مدلسازی یکپارچه دو نمونه از زبان تصویری استاندارد هستند که به تسهیل انجام کارها با ترویج قانونمندی، حذف ابهام و استفاده از حمایت ابزار نرمافزاری برای تبادل اطلاعات پیچیده کمک میکنند. درواقع زیستشناسی بااینکه دارای بیشترین حجم اطلاعات نموداری نسبت به اطلاعات نوشتاری است همچنان فاقد نمادهای گرافیکی استاندارد است. سیل اخیر دانش زیستشناسی این نقص را موردتوجه بیشتری قرار داده است. برای نیل به این هدف ما به ارائه سیستم نمادهای گرافیکی زیستشناسی (SBGN) میپردازیم، یکزبان تصویری که توسط جامعه بیوشیمی، طراحان و دانشمندان علوم رایانه فراهمشده است.SBGN شامل سه زبان تکمیلی است: نمودار فرایند، نمودار رابطه موجود و نمودار جریان فعالیت. این سه مورد به دانشمندان این امکان را میدهد که شبکهای از تراکنشهای شیمیایی را درروشی استاندارد و بدون ابهام نشان دهند. ما معتقدیم که SBGN یک نمایش درست و مؤثر، تجسم، ذخیرهسازی، تبادل و استفاده مجدد از اطلاعات را در تمامی انواع دانش زیستشناسی از تنظیم ژن گرفته تا متابولیسم و علائم سلولی را ترویج میکند.
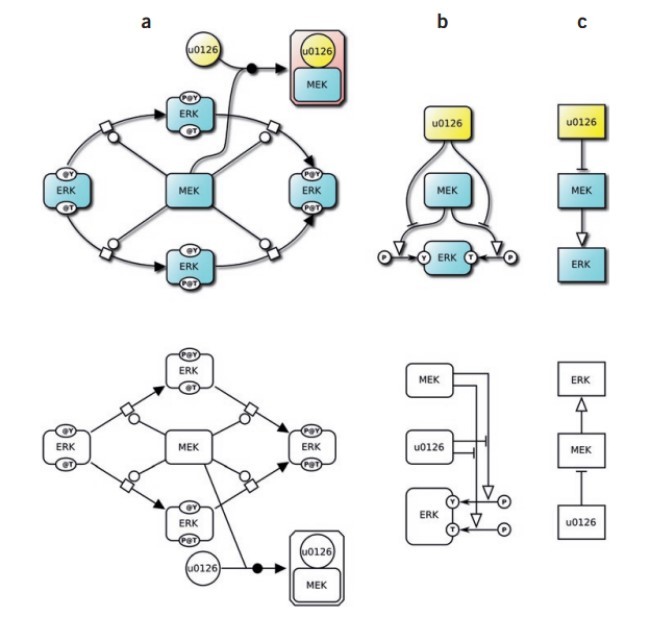
ناپلئون بناپارت میگوید یک طرح خوب بهتر از یک سخنرانی طولانی است. چنین ادعایی مصداق خود را جایی بهتر از تصاویر فنی نشان نخواهد داد. نمودارها در ماهیت ریشه در قوای شناختی ذاتی ما دارند که انسانها از زمانی پیش از نگارههای نیاکانمان در غارها نیز دارای آن بودهاند. جای شگفتی نیست که ما در هر زمینهای که تلاش میکنیم بازهم به سراغ نمودارها میرویم. این قضیه در مورد زبانهای نوشتاری انسان نیز صدق میکند، ارتباط برقرار کردن شامل نگارههایی است که لازمهاش آن است که هم نویسنده و هم خواننده درباره نمادها، قوانین ترتیب قرار گرفتنشان و تفسیر نتایج باهم به توافق رسیده باشند. ایجاد و استفاده گسترده از نمادهای استاندارد به بسیاری از رشتهها امکان داده که پیشرفت کنند. بهسختی میتوان تصور کرد که صنعت الکترونیک امروزی با ابزارهای خودکار و قدرتمندش که بر اساس تصویر طراحیشدهاند فاقد نمادهای استاندارد اولیه در نمودارهای مداری باشد. چنین چیزی در زیستشناسی اهمیتی نداشت. در زیستشناسی برخلاف ماهیت تصویری بیشتر اطلاعات تبادلی، این رشته هم چنان تحت نفوذ نمادهای گرافیکی تک موردی بود که در میان پژوهشگران، انتشارات، کتابها و برنامههای نرمافزاری نقاط اشتراک کمی داشتند. هیچ زبان تصویری استانداردی برای توصیف شبکههای تراکنش بیو شیمیایی، علائم درون ـ و برون سلولی، تنظیم ژنها ـ مفاهیمی که هسته مرکزی پژوهشهای امروزی درزمینه مولکولی، سیستمها و ترکیب زیستی هستند، وجود نداشت. درواقع، نزدیکترین نماد به شکل استاندارد که مدتهای طولانی در بسیاری از نقشههای مسیریابی علائم و متابولیک استفاده میشد، فاقد انسجام بین منابع بود و شامل ابهامات نامطلوبی نیز بود (شکل 1). بهعلاوه این نمایشهای تجربی موجود بااینکه خوشساخت بودند اما مبهم بودند و فقط برای نیازهای خاص مناسب بودند مانند نشان دادن شبکه متابولیک یا مسیرهای علائم یا تنظیم ژنها.
عصر زیستشناسی مولکولی، و اخیراً با ظهور ژنومی و سایر فناوریهای توان ـ بالا منجر به افزایش سرسامآور دادههایی شده است که نیاز به تفسیر دارند. استفاده از نرمافزارها برای کمک به فرمولبندی فرضیهها، طراحی آزمایشها و تفسیر نتایج نیز مطلوب همه است. ما بهعنوان گروهی از جامعه بیوشیمی، طراحان و دانشمندان رایانه که بر روی سیستمهای زیستی کار میکنند معتقدیم که ایجاد نمادهای گرافیکی استاندارد گامی مهم در جهت انتقال صحیح و مؤثر دانش زیستشناسی در بین جوامع مختلف خواهد بود. برای نیل به این منظور ما پروژه SBGN را از سال 2005 باهدف توسعه و استانداردسازی نمادهای گرافیکی نظاممند و روشن برای کاربرد در سیستمهای زیستی و مولکولی آغاز کردیم.
پیشینه تاریخی
نمایش گرافیکی فرایندهای سلولی و بیو شیمیایی در متون بیو شیمی به شصت سال پیش بازمیگردد که نقطه اوجش در نمودارهایی بود که نیکلسون و میکال با دست روی دیوار میکشیدند. آن نگارهها به توصیف فرایندهای تغییر مجموعهای از ورودیها به مجموعهای از خروجیها میپرداختند درواقع نمودارهای فرایند یا انتقال حالت بودند. این سبک از نمودارها در اولین سیستمهای پایگاه دادهای که به شرح شبکههای متابولیک میپرداختند، تقلید شدند.این نمودارهای تقلیدی شاملEMP^6، EcoCyc^7 وKEGG^8 بودند. نمادهای بیشتری نیز در ابزارهای نرمافزاری ویژه بسته به حسن اجراییشان مانند طراحان شبکه و مسیریابی تعریف شدند (برای مثال ، NetBuilder، Patika ،JDesigner، CellDesigner). آن نمادهای گرافیکی استاندارد نبودند و درک آنها بیشتر بر اساس مثالهای مرتبط بادانش موجود از فرایندهای بیوشیمیایی بود. بااینکه نگارههای کلاسیک بهدرستی اطلاعات بیو شیمیایی را منتقل میکردند، زمانی که بحث به ژنوم یا زیست مولکولی میرسید به انواع دیگر نمودارها برای نمایش مسیرهای علائم و اطلاعات غیرمستقیم و ناقص نیاز بود. آن نمودارها بیشتر تقلیدی از نمادهای تجربی استفادهشده توسط زیست شناسان بود که به توصیف روابط بین عناصر یا جریان فعالیت یا تأثیر میپرداختند. بعدازآن فهرستی از تصویر نگارههای استاندارد (جعبه 1) برای نمایش مفاهیم مشخص فراهم شد. تلاشها برای ارائه یک طرح تعریفشده مشخص ابتدا توسط Kurt Kohn با نقشه واکنشهای مولکولیاش (MIM) آغاز شد، که نهتنها به تعریف مجموعهای از نمادها میپرداخت بلکه به تشریح واکنشها و روابط مولکولها نیز میپرداخت. نماد MIM بر سایر پیشنهادهای پژوهشی نیز تأثیر گذاشت. برخی از پژوهشها به ادامه توصیف نمودارهای فرایند پرداختند و این کار را نهتنها با نمادهای استاندارد انجام دادند بلکه دستور زبان ویژه آن را نیز تعریف کردند.
پروژه SBGN
علیرغم محبوبیتی که برخی از کارهای تحقیقاتی فوق داشتند هیچیک از نمادهای استفادهشده فرم استانداردی به خود نگرفتند. بخشی از آن به این دلیل بود که این پژوهشها تا جایی پیش رفتند که نمادی را پیشنهاد داده باشند یا آن را دریک نرمافزار اجرایی کنند. برخی از ما در توسعه سیستمهای زبان نشانهگذاری زیستی (SBML) مشارکت داشتیم، ما در آن پروژه آموختیم که ایجاد یک استاندارد بدون تلاشی روشن و عینی با در نظر داشتن جامعه دانشمندان و نیز اتفاق آرا در میان آنان کاری بس دشوار خواهد بود. ما پروژه SBGN را با این ذهنیت سازماندهی کردیم.
برای اینکه پروژه SBGN موفقیتآمیز باشد باید نیازهای فنی و عملی را برآورده میکرد و در جامعه متنوع و گوناگون زیست شناسان، زیستشیمیدانها، بیو انفورماتیکها، متخصصان علوم ژنتیک، نظریهپردازان و مهندسان نرمافزار نیز با آغوش باز پذیرفته میشد. در مراحل اولیه پیشینه تاریخی ما به دنبال ایجاد اصول فراگیری بودیم تا SBGN را برای نیل به اهدافی که به ترتیب اولویت رتبهبندی شدهاند بهپیش براند.
نماد میبایست.
• فارغ از قیدوبندهای هوشمندانه باشد تا جامعه علمی بتواند آزادانه از آن استفاده کند؛
• ازلحاظ دستوری و معنایی دارای انسجام و فاقد هرگونه ابهام باشد؛
• از نمایههای زیستی رایج و گوناگون و نیز از ویژگیها و تراکنشهای آنان پشتیبانی کند؛
• تعداد نمادها و معانی آنها را به حداقل میزان لازم برساند تا زمینه درک و یادگیری آنها را فراهم کند؛
• ازلحاظ تصاویر منسجم و دقیق باشد، از نمادهای تشخیص پذیر استفاده کند؛
• از مقیاس پشتیبانی کرده تا بتواند از عهده اندازه نمودار و پیچیدگیان برآید؛
• از تنظیم اتوماتیک نمودارها بهوسیله نرمافزارهایی که بر اساس مدلهای ریاضی طراحیشدهاند، پشتیبانی کند.
بسیاری از اصول طراحی که در قسمتهای فوق به آنها اشاره شد با پژوهش بر روی زبانهای تصویری تشدید شدهاند و نیز همراه با آن مطالعاتی که هدفشان درک نیازهای غایی کاربر در مسیرهای تصویرسازی بوده است، ولی بااینحال ما آنها را از مجموعه تجارب در دسترسمان با توسعه نمادها و نرمافزار به دست آوردهایم. علاوه بر اصولی که ذکر شدند ما بران بودیم تا از بسیاری مسائل و مشکلات (جدول 1) که بر برخی نمادهای موجود تأثیر میگذاشت اجتناب کنیم.
Circuit diagrams and Unified Modeling Language diagrams are just two examples of standard visual languages that help accelerate work by promoting regularity, removing ambiguity and enabling software tool support for communication of complex information. Ironically, despite having one of the highest ratios of graphical to textual information, biology still lacks standard graphical notations. The recent deluge of biological knowledge makes addressing this deficit a pressing concern. Toward this goal, we present the Systems Biology Graphical Notation (SBGN), a visual language developed by a community of biochemists, modelers and computer scientists. SBGN consists of three complementary languages: process diagram, entity relationship diagram and activity flow diagram. Together they enable scientists to represent networks of biochemical interactions in a standard, unambiguous way. We believe that SBGN will foster efficient and accurate representation, visualization, storage, exchange and reuse of information on all kinds of biological knowledge, from gene regulation, to metabolism, to cellular signaling.
“Un bon croquis vaut mieux qu’un long discours” (“A good sketch is better than a long speech”), said Napoleon Bonaparte. This claim is nowhere as true as for technical illustrations. Diagrams naturally engage innate cognitive faculties1 that humans have possessed since before the time of our cave-drawing ancestors. Little wonder that we find ourselves turning to them in every field of endeavor. Just as with written human languages, communication involving diagrams requires that authors and readers agree on symbols, the rules for arranging them and the interpretation of the results. The establishment and widespread use of standard notations have permitted many fields to thrive. One can hardly imagine today’s electronics industry, with its powerful, visually oriented design and automation tools, without having first established standard notations for circuit diagrams. Such was not the case in biology2. Despite the visual nature of much of the information exchange, the field was permeated with ad hoc graphical notations having little in common between different researchers, publications, textbooks and software tools. No standard visual language existed for describing biochemical interaction networks, inter- and intracellular signaling gene regulation—concepts at the core of much of today’s research in molecular, systems and synthetic biology. The closest to a standard is the notation long used in many metabolic and signaling pathway maps, but in reality, even that lacks uniformity between sources and suffers from undesirable ambiguities (Fig. 1). Moreover, the existing tentative representations, however well crafted, were ambiguous, and only suitable for specific needs, such as representing metabolic networks or signaling pathways or gene regulation.
The molecular biology era, and more recently the rise of genomics and other high-throughput technologies, have brought a staggering increase in data to be interpreted. It also favored the routine use of software to help formulate hypotheses, design experiments and interpret results. As a group of biochemists, modelers and computer scientists working in systems biology, we believe establishing standard graphical notations is an important step toward more efficient and accurate transmission of biological knowledge among our different communities. Toward this goal, we initiated the SBGN project in 2005, with the aim of developing and standardizing a systematic and unambiguous graphical notation for applications in molecular and systems biology.
Historical antecedents
Graphical representation of biochemical and cellular processes has been used in biochemical textbooks as far back as sixty years ago3, reaching an apex in the wall charts hand drawn by Nicholson4 and Michal5. Those graphs describe the processes that transform a set of inputs into a set of outputs, in effect being process, or state transition, diagrams. This style was emulated in the first database systems that depicted metabolic networks, including EMP6, EcoCyc7 and KEGG8. More notations have been ‘defined’ by virtue of their implementation in specialized software tools such as pathway and network designers (e.g., NetBuilder9, Patika10, JDesigner11, CellDesigner12). Those graphical notations were not standardized, and their understanding relied mainly on relating examples with one’s preexisting knowledge of biochemical processes. Although the classical graphs adequately conveyed information about biochemistry, other types of diagrams were needed to represent signaling pathways, and incomplete or indirect information, as coming from molecular biology or genomics. Those conventions effectively mimicked the empirical notations used by biologists, describing either the relationships between elements13,14 or the flow of activity or influence15–17. Lists of standard glyphs (Box 1) to represent identified concepts were then provided. The efforts to create rigidly defined schema were pioneered by Kurt Kohn with his Molecular Interaction Maps (MIM), which defined not only a set of symbols but also a syntax to describe interactions and relationships of molecules18,19. The MIM notation influenced other proposals14. Several proposals followed to describe process diagrams, not only with standard symbols but also defined grammars20–23.
The SBGN project
Despite the popularity of some of the efforts mentioned above, none of the notations has acquired the status of a community standard. This can be attributed partly to the fact that the efforts only went as far as to propose notations, or implement them in software. Several of us have been involved in the development of the Systems Biology Markup Language (SBML)24, from which we learned that establishing a standard is extremely difficult without an explicit, concerted, effort to engage a community and build a consensus among participants. We organized the SBGN project with this lesson in mind.
For SBGN to be successful, it must satisfy a majority of technical and practical needs and be embraced by a diverse community of biologists, biochemists, bioinformaticians, geneticists, theoreticians and software engineers. Early in the project’s history, we established the following overarching principles to help steer SBGN toward those aims, ranked by rough hierarchical order of precedence.
The notation should
• be free of intellectual property restrictions to allow free use by the community;
• be syntactically and semantically consistent and unambiguous;
• support representation of diverse common biological objects, their properties and their interactions;
• keep the number of symbols and syntax to a minimum to help comprehension and learning by humans;
• be visually consistent and concise, using discriminable symbols;
• support modularity to help cope with diagram size and complexity;
• support the automated generation of diagrams by software starting from mathematical models.
Many of the design principles above resonate with research on visual languages25,26 and studies aimed at understanding end-user needs in pathway visualization27, although we derived them from our collective hands-on experiences with developing notations and software. In addition to these principles, we also sought to avoid many problems (Table 1) that affect some existing notations.
پیشینه تاریخی
پروژه SBGN
سه زبان SBGN
نمودار فرایند SBGN
نمودار رابطه موجود SBGN
نمودار جریان فعالیت SBGN
مشارکت و چشماندازهای آینده
منابع
Historical antecedents
The SBGN project
The three languages of SBGN
SBGN process diagram
SBGN entity relationship diagram
SBGN activity flow diagram
Participation and future prospects
References
